algoritmul de bază local Alignment Search Tool (BLAST) se află în centrul unei suite gratuite de resurse online disponibile prin Centrul Național pentru Informații Biotehnologice (NCBI). În timp ce majoritatea cercetătorilor sunt conștienți de BLAST ca instrument de aliniere a secvențelor, suita BLAST NCBI oferă mult mai mult! Voi acoperi în profunzime modul de utilizare a acestor resurse pentru a localiza polimorfisme nucleotidice unice (SNP) într-o genă; proiectarea primerilor cu primer-BLAST; și validarea țintelor primer.
sfat unu: Cum să găsiți SNPs
având în vedere importanța SNPs atât în boală, cât și în cercetare, NCBI oferă instrumente pentru colaționarea SNPs-urilor raportate de o genă. Pentru a găsi SNP-uri, începeți de la pagina principală a NCBI și introduceți gena de interes în bara de căutare. Selectați SNP din meniul derulant toate bazele de date din stânga barei de căutare, așa cum se arată mai jos:
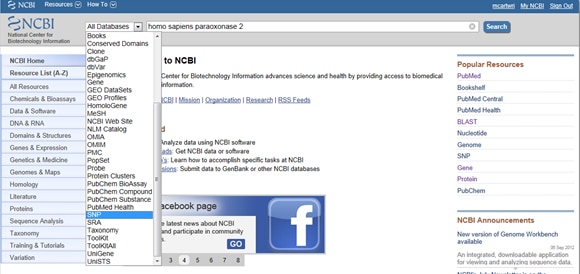
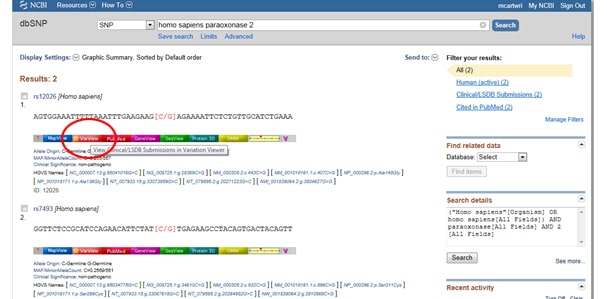
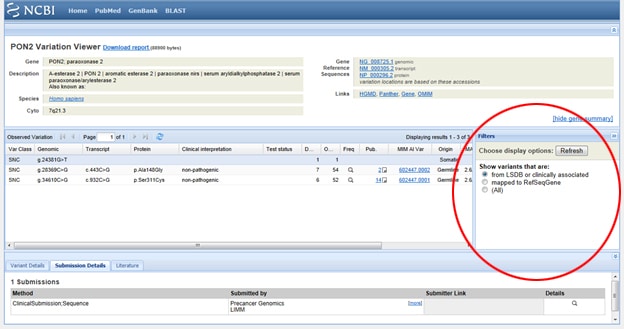
trucul unu: trebuie să filtrați rezultatele astfel încât să vă uitați doar la rezultatele asociate clinic? Accesați caseta de afișare etichetată filtre din dreapta listei SNP sub variație observată. După ce ați selectat opțiunea de filtrare, asigurați-vă că apăsați butonul Reîmprospătare.
sfat doi: Cum să proiectați primeri
NCBI oferă Primer-BLAST pentru proiectarea automată a primerilor pe baza unei secvențe de interogare. Pentru a începe proiectarea primerilor, accesați pagina principală BLAST și derulați în jos până la opțiunea Primer-BLAST sub explozie specializată. Introduceți secvența țintă fie prin tăiere și lipire, fie, dacă este listată în bazele de date ale NCBI, ca număr de aderare. Am acoperi unele opțiuni de personalizare de mai jos, dar în acest moment, puteți genera primeri fără a face nici o personalizare suplimentare!
interval: în partea dreaptă a casetei pentru introducerea secvenței dvs., puteți specifica intervalul exact (numerotat de la 5′ la 3′, de la începutul secvenței dvs.) al țintei care va fi luată în considerare pentru proiectarea primerilor înainte și înapoi.
utilizați propriul primer forward (5′->3′ pe Plus strand): selectați acest lucru dacă v-ați proiectat deja grundurile și doriți ca Primer-BLAST să ofere câteva analize (de exemplu, Tm) despre ele.
dimensiunea produsului PCR: setați aici gama de lungimi acceptabile ale produselor PCR.
# de primeri de returnat: aceasta stabilește numărul preferat de seturi candidate de primeri de luat în considerare. Rețineți că nu este o garanție, mai ales dacă parametrii dvs. sunt prea stricți sau fără sens (de exemplu, ați specificat un produs sub dimensiunea produsului PCR care nu poate fi mai mare de 500 bp, dar sub interval doriți doar să luați în considerare grundurile la mai mult de 1 kb distanță).
temperatura de topire a grundului: Acest lucru vă permite să specificați Tm (pentru o reîmprospătare rapidă a temperaturii de topire, consultați sfaturile noastre pentru qPCR și proiectarea regulată a primerului PCR).
Exon junction span: dacă doriți să excludeți ADN-ul genomic (unde exonii sunt împărțiți la introni necodificatori), atunci setați acest lucru la Primer trebuie să se întindă pe o joncțiune exon-exon.
verificarea specificității: dacă nu doriți ca Primer-BLAST să returneze primerii care vor ieși din țintă (în general nu este recomandat!), lăsați acest lucru verificat și specificați organismul din care provin probele dvs., precum și ce Bază de date să utilizați, în funcție de dacă vizați ARNm, gDNA etc. Prin activarea verificării specificității, Primer-BLAST va exclude primerii care ar putea amplifica ceva în afara secvenței dvs. țintă.
manipularea variantei de îmbinare: dacă selectați această opțiune – fezabilă numai dacă lucrați la secvențe de ARNm – atunci Primer-BLAST nu va exclude perechile de grunduri care ar putea amplifica mai multe variante de îmbinare a ARNm ale țintei dvs. Acest lucru nu înseamnă, totuși, că vă va oferi perechi de grunduri care cuprind toate variantele de îmbinare cunoscute! Pur și simplu îți slăbești criteriile țintă.
după ce ați introdus secvența și ați personalizat după cum este necesar, derulați în jos până la partea de jos a paginii și, după verificare utilizați vizualizarea grafică nouă, apăsați Obțineți primeri. Aceasta va returna o hartă a locului în care perechile de grunduri sugerate vă vor amplifica ținta, precum și analize ale primerilor: lungimea lor, locația exactă, Tm-urile respective, GC% și scorurile care reflectă auto-complementaritatea (cu 0,00 care nu reflectă nicio completare prezisă).
sfat trei: Cum de a prezice obiective Primer
cum puteți verifica dacă primeri dvs. a lovit ceva off-țintă? Du-te la Primer-BLAST. În caseta de interogare, introduceți primerul înainte (5′ la 3′). Acum introduceți 20 N într-un rând pentru a separa grundurile în alinieri individuale, care nu se suprapun. După N ‘S, introduceți grundul invers (de asemenea, 5′ la 3’), așa cum se arată mai jos:
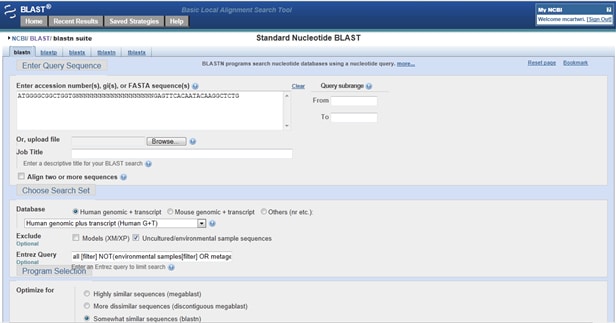
odată ce obțineți rezultatele, verificați-le pentru anumite combinații. Dacă grundul dvs. înainte se aliniază pe firul înainte (firul adnotat Plus/Plus) și grundul invers se aliniază la aceeași lovitură, dar pe firul invers (firul Plus/Minus), atunci grundurile dvs. pot amplifica acea lovitură.
trucul doi: rezultatele dvs. includ lucruri puțin probabil să vă fi contaminat probele PCR, cum ar fi babuinii de măsline și neanderthalienii? Dacă lucrați cu eșantioane umane sau de șoarece, asigurați-vă că aveți cele specificate în baza de date. Alternativ, puteți exclude anumite specii.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
v-a ajutat acest lucru? Apoi, vă rugăm să partajați cu rețeaua.