het basisalgoritme voor Local Alignment Search (BLAST) vormt de kern van een gratis reeks online bronnen die beschikbaar zijn via het National Center for Biotechnology Information (NCBI). Terwijl de meeste onderzoekers zich bewust zijn van BLAST als een sequence alignment tool, biedt NCBI ‘ s BLAST suite zoveel meer! Ik zal dieper ingaan op het gebruik van deze middelen om enkelvoudige nucleotide polymorfismen (SNP ‘ s) in een gen te lokaliseren; het ontwerpen van primers met Primer-BLAST; en het valideren van primer targets.
Tip één: Hoe SNP ‘s
te vinden gezien het belang van SNP’ s in zowel ziekte ALS onderzoek, biedt NCBI hulpmiddelen voor het verzamelen van de gerapporteerde SNP ‘ s van een gen. Om SNPs te vinden, beginnen bij NCBI ‘ s homepage en typ uw gen van belang in de zoekbalk. Selecteer SNP in het keuzemenu alle Databases links van de zoekbalk, zoals hieronder weergegeven:
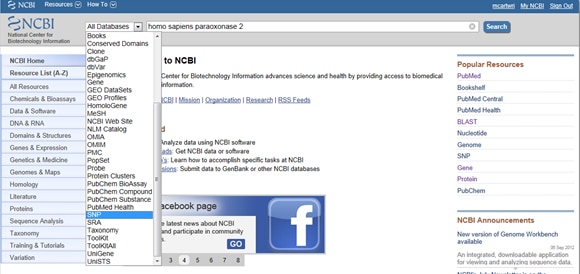
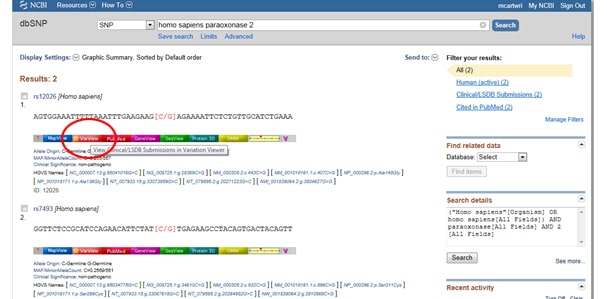
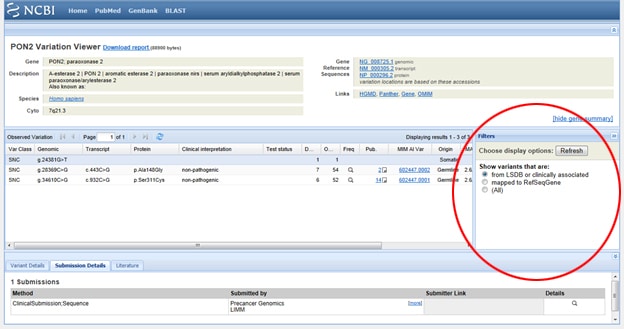
Truc één: moet u de resultaten filteren zodat u alleen naar klinisch geassocieerde resultaten kijkt? Ga naar het venster Filters aan de rechterkant van de SNP-lijst onder waargenomen variatie. Nadat u uw filteroptie hebt geselecteerd, moet u op de knop Vernieuwen drukken.
Tip twee: Hoe Primers te ontwerpen
NCBI biedt Primer-BLAST Voor het automatisch ontwerpen van primers op basis van een query-sequentie. Om te beginnen met het ontwerpen van primers, ga naar de BLAST homepage en scroll naar beneden naar de Primer-BLAST optie onder gespecialiseerde BLAST. Voer uw doelreeks in door te knippen en te plakken of, als het in NCBI ‘ s databases wordt vermeld, als een toetredingsnummer. Ik heb betrekking op een aantal aanpassingsopties hieronder, maar op dit punt, kunt u primers genereren zonder enige extra aanpassing!
Range: aan de rechterkant van het vakje voor het invoeren van uw reeks, kunt u het exacte bereik opgeven (als genummerd 5′ tot 3′, vanaf het begin van uw reeks) van het doel dat zal worden overwogen voor het ontwerpen van de vooruit en achteruit primers.
gebruik mijn eigen forward primer (5′->3′ op plus strand): Selecteer dit als u uw primers al hebt ontworpen en wilt dat Primer-BLAST een aantal analytics (bijvoorbeeld Tm) over hen levert.
PCR-productgrootte: Stel hier het bereik van aanvaardbare lengtes van de PCR-producten in.
# van de te retourneren primers: dit stelt het gewenste aantal kandidaat – sets primers in om te overwegen. Merk op dat het geen garantie is, vooral als je parameters te streng of onzinnig zijn (bijvoorbeeld je hebt een product opgegeven onder PCR-productgrootte die niet meer dan 500 bp kan zijn, maar onder bereik wil je alleen primers beschouwen die meer dan 1 kb uit elkaar liggen).
smelttemperatuur van de Primer: Hiermee kunt u uw Tm specificeren (voor een snelle opfriscursus op smelttemperatuur, Bekijk onze tips voor qPCR en regular PCR primer design).
exon junction span: als u genomisch DNA wilt uitsluiten (waar exonen worden gedeeld door niet-coderende introns) dan zet dit op Primer moet een exon-exon junction overspannen.
Specificiteitscontrole: tenzij u wilt dat Primer-BLAST primers retourneert die off-target gaan (over het algemeen niet aanbevolen!), laat dit gecontroleerd en specificeer het organisme uw steekproeven komen van evenals welk gegevensbestand te gebruiken, afhankelijk van als u zich richt op mRNA, gDNA, enz. Door de specificiteitscontrole in te schakelen, zal Primer-BLAST primers uitsluiten die iets buiten je doelreeks kunnen versterken.
Splice variant handling: Als u deze optie selecteert – alleen mogelijk als u werkt met mRNA sequenties-dan zal Primer-BLAST primerparen niet uitsluiten die meerdere mRNA splice varianten van uw doel kunnen versterken. Dit betekent echter niet dat het u primer paren die alle bekende splice varianten omvatten zal geven! Je maakt gewoon je doelcriteria los.
zodra u uw reeks hebt ingevoerd en naar behoefte aangepast, scroll naar beneden naar de onderkant van de pagina en, na het controleren Gebruik nieuwe grafische weergave, druk op Get Primers. Dit zal een kaart van waar de voorgestelde primer paren zal uw doel te versterken, evenals analytics op de primers: hun lengte, precieze locatie, respectieve Tm ‘ s, GC%, en scores die zelf-complementariteit (met 0,00 als gevolg van geen voorspelde aanvulling).
Tip drie: Hoe voorspel je Primer Targets
hoe kun je controleren of je primers iets buiten het doel raken? Ga naar Primer-BLAST. Voer in het vak query uw Forward primer (5′ to 3′) in. Typ nu 20 N ‘ s in een rij om de primers te scheiden in individuele, niet-overlappende uitlijningen. Voer na de N ‘S uw reverse primer in (ook 5′ tot 3’), zoals hieronder getoond:
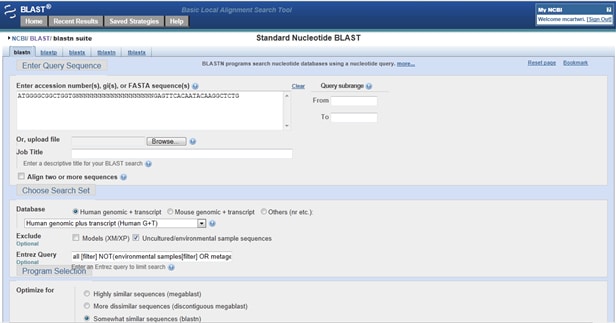
zodra u uw resultaten, controleer ze voor bepaalde combinaties. Als je forward primer uitgelijnd is op de forward streng (geannoteerde streng Plus / Plus) en je reverse primer uitgelijnd is op dezelfde hit, maar op de reverse streng (streng Plus/Minus), dan kunnen je primers die hit versterken.
Truc twee: bevatten uw resultaten dingen die waarschijnlijk uw PCR-monsters hebben verontreinigd, zoals olijfbavianen en Neanderthalers? Als u met mens-of muismonsters werkt, zorg er dan voor dat u deze hebt opgegeven onder Database. Als alternatief kunt u specifieke soorten uitsluiten.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
heeft dit u geholpen? Dan kunt u delen met uw netwerk.