Basic Local Alignment Search Tool (BLAST) algoritmen er i hjertet av en gratis pakke med elektroniske ressurser tilgjengelig gjennom National Center For Biotechnology Information (NCBI). MENS de fleste forskere er klar OVER BLAST som et sekvensjusteringsverktøy, TILBYR NCBIS BLAST suite så mye mer! Jeg vil dekke grundig hvordan du bruker disse ressursene til å finne single nucleotid polymorphisms (SNPs) i et gen; design primere Med Primer-BLAST; og validere primer mål.
Tips En: HVORDAN finne SNPs
GITT betydningen Av SNPs i både sykdom og forskning, GIR NCBI verktøy for å sortere et gens rapporterte SNPs. For å finne SNPs, start PÅ NCBIS hjemmeside og skriv inn ditt gen av interesse i søkefeltet. Velg SNP fra rullegardinmenyen alle Databaser til venstre for søkefeltet, som vist nedenfor:
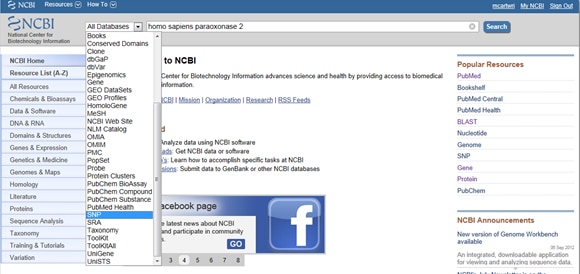
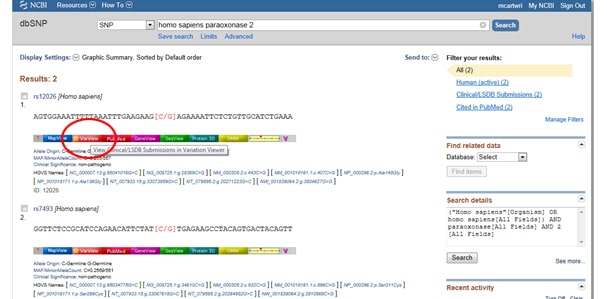
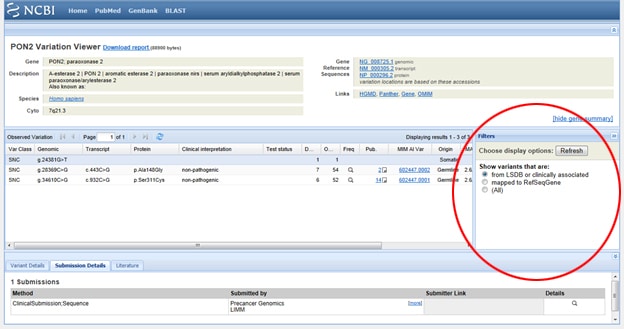
TIPS To: Hvordan Designe Primere
NCBI gir Primer-BLAST for automatisk utforming av primere basert på en spørringssekvens. For å begynne å designe primere, gå TIL BLAST hjemmeside og bla ned Til Primer-BLAST alternativet Under Specialized BLAST. Skriv inn målet sekvens enten ved cut-and-paste eller, hvis det er oppført I NCBI databaser, som en tiltredelse nummer. Jeg dekker noen tilpasningsalternativer nedenfor, men på dette punktet kan du generere primere uten å gjøre noen ekstra tilpasning!
Område: til høyre for boksen for å skrive inn sekvensen din, kan du angi det eksakte området (som nummerert 5′ til 3′, fra starten av sekvensen din) av målet som vil bli vurdert for å designe forover og bakover primere.
Bruk min egen forward primer (5 ‘- > 3 ‘ på pluss strand): Velg dette hvis Du allerede har designet primere og Vil At Primer-BLAST skal gi noen analyser (f. eks.
PCR produktstørrelse: Angi rekkevidden av akseptable lengder AV PCR-produktene her.
# av primere å returnere: dette setter ditt foretrukne antall kandidatsett med primere å vurdere. Merk at det ikke er en garanti, spesielt hvis parametrene dine er for strenge eller nonsensiske(f. eks. du angav et produkt under PCR – produktstørrelse som ikke kan være mer enn 500 bp, men Under Rekkevidde vil du bare vurdere primere mer enn 1 kb fra hverandre).
Primer smeltetemperaturer: Dette lar deg spesifisere Din Tm (for en rask oppfriskning på smeltetemperatur, sjekk ut våre tips for qPCR og vanlig PCR primer design).
Exon junction span: hvis du vil ekskludere genomisk DNA (hvor eksoner er delt av ikke-kodende introner), så sett Dette Til Primer må span en exon-exon junction.Spesifisitet sjekk: Med mindre Du Vil Primer-BLAST å returnere primere som vil gå off-target (vanligvis ikke anbefalt!), la dette kontrolleres og spesifiser Organismen dine prøver kommer fra, samt hvilken Database som skal brukes, avhengig av om du målretter mot mRNA, gDNA, etc. Ved å aktivere spesifisitet sjekk, Primer-BLAST vil utelukke primere som kan forsterke noe utenfor målet sekvens.
Splice variant håndtering: hvis du velger dette alternativet-bare mulig hvis du arbeider ut av mRNA sekvenser – Da Primer-BLAST vil ikke utelukke primer par som kan forsterke flere mRNA splice varianter av målet. Dette betyr imidlertid ikke at det vil gi deg primerpar som omfatter alle kjente spleisevarianter! Du løsner bare målkriteriene dine.
når du har skrevet inn sekvensen din og tilpasset etter behov, bla ned til bunnen av siden, og etter å ha sjekket Bruk ny grafisk visning, trykk Få Primere. Dette vil returnere et kart over hvor de foreslåtte primerparene vil forsterke målet ditt, samt analyser på primerne: deres lengde, presis plassering, respektive Tm-er, GC% og score som reflekterer selvkomplementaritet(med 0,00 som reflekterer ingen forventet komplement).
Tips Tre: Hvordan Forutsi Primer Mål
Hvordan kan du sjekke om primere treffer noe off-target? Gå Til Primer-BLAST. I spørringsboksen, skriv inn din fremover primer (5 ’til 3′). Skriv inn 20 N på rad for å skille primerne i individuelle, ikke-overlappende justeringer. Etter N, skriv inn omvendt primer (også 5 ’til 3′), som vist nedenfor:
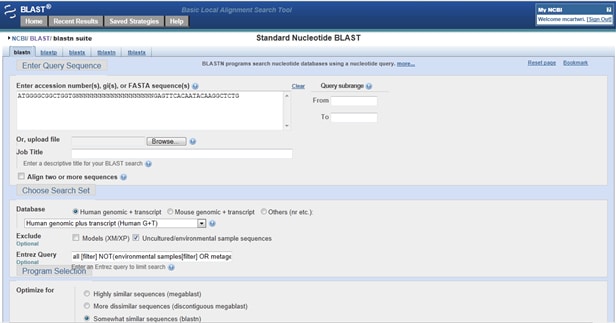
når du får resultatene dine, sjekk dem for bestemte kombinasjoner. Hvis fremoverprimeren justerer seg på fremoverstrengen (annotert Streng Pluss / Pluss) og omvendt primer justerer seg til samme treff, men på omvendt streng (Streng Pluss / Minus), kan primerne forsterke det treffet.Trick Two: inkluderer resultatene dine ting som er usannsynlig å ha forurenset PCR-prøvene dine, for eksempel olivenbavianer og Neandertalere? Hvis du arbeider med menneske-eller museprøver, må du sørge for at du har de som er angitt Under Database. Alternativt kan du utelukke bestemte arter.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
har dette hjulpet deg? Vennligst del med nettverket ditt.