基本的なローカルアライメント検索ツール(BLAST)アルゴリズムは、国立バイオテクノロジー情報センター(NCBI)を介して利用可能なオンラインリソースの無料スイートの中心にあります。 ほとんどの研究者は、シーケンスアライメントツールとしてBLASTを認識していますが、NCBIのBLAST suiteはそんなに多くを提供しています! これらのリソースを使用して、遺伝子内の一塩基多型(Snp)を特定する方法、Primer-BLASTを使用してプライマーを設計する方法、プライマーターゲットを検証する方法を
ヒント1: SNPsを見つける方法
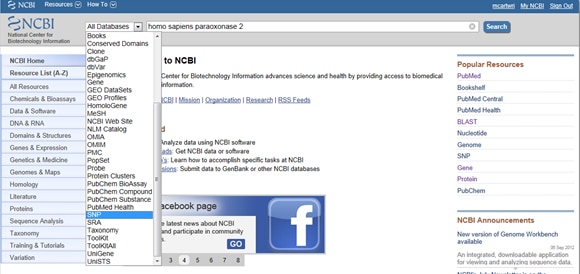
疾患と研究の両方におけるSNPsの重要性を考えると、NCBIは遺伝子の報告されたSNPsを照合するためのツールを提供します。 Snpを検索するには、NCBIのホームページから開始し、検索バーに関心のある遺伝子を入力します。 以下に示すように、検索バーの左側にあるすべてのデータベースドロップダウンメニューからSNPを選択します。
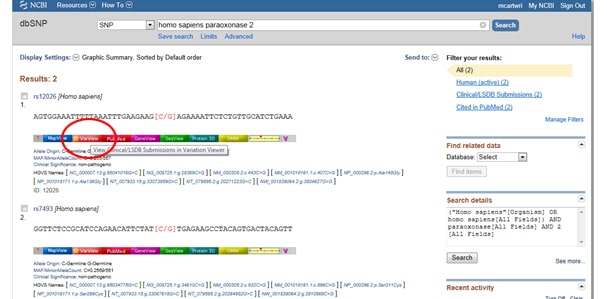
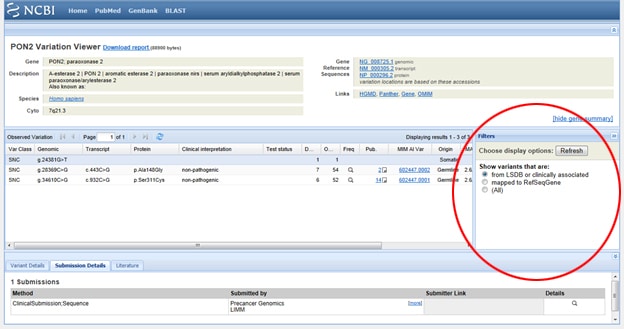
トリックワン:あなただけの臨床的に関連する結果を見ているように、結果をフィルタリングする必要がありますか? SNPリストの右側にある”観測された変動”の下にある”フィルタ”というラベルの表示ボックスに移動します。 フィルタオプションを選択したら、必ず[更新]ボタンを押してください。
ヒント二つ:プライマーを設計する方法
NCBIは、クエリシーケンスに基づいてプライマーを自動的に設計するためのPrimer-BLASTを提供しています。 プライマーの設計を開始するには、BLASTホームページに移動し、Specialized BLASTの下のPrimer-BLASTオプションまでスクロールダウンします。 カットアンドペーストでターゲットシーケンスを入力するか、NCBIのデータベースに記載されている場合はアクセッション番号として入力します。 私は以下のいくつかのカスタマイズオプションをカバーしていますが、この時点で、追加のカスタマイズを行うことなく、プライマーを生成することができます!
Range:シーケンスを入力するためのボックスの右側に、順方向および逆方向のプライマーを設計するために考慮されるターゲットの正確な範囲(シーケンスの自分のフォワードプライマーを使用します(5′->3’on plus strand):すでにプライマーを設計していて、Primer-BLASTにそれらについての分析(Tmなど)を提供したい場合は、これPCR製品サイズ:PCR製品の許容可能な長さの範囲をここに設定します。
返すプライマーの数:これは、考慮するプライマーの候補セットのお好みの数を設定します。 特に、パラメータが厳密すぎるか無意味な場合(たとえば、500bpを超えることはできませんが、範囲の下ではプライマーを1kb以上離れて検討したいだけのpcr product sizeの下で製品を指定した場合)、それは保証ではないことに注意してください。
プライマーの溶融温度: これはあなたのTmを指定することを可能にする(溶ける温度の速い復習のために、qPCRおよび規則的なPCRのプライマー設計のための私達の先端を点検し
エクソン接合スパン:あなたは(エクソンが非コードイントロンで分割されている)ゲノムDNAを除外したい場合は、プライマーは、エクソン-エクソン接合特異性チェック:Primer-BLASTがオフターゲットになるプライマーを返すようにしたい場合を除きます(一般的にはお勧めしません!)、これをチェックしたままにして、mRNA、gDNAなどをターゲットにしているかどうかに応じて、サンプルがどこから来ているか、使用するデータベースを指定します。 特異性チェックを有効にすることにより、Primer-BLASTは、ターゲット配列の外に何かを増幅する可能性のあるプライマーを除外します。
スプライスバリアントの処理:このオプションを選択した場合–mRNA配列を操作している場合にのみ実行可能です–Primer-BLASTは、ターゲットの複数のmRNAスプライスバリアントを増幅する可能性のあるプライマーペアを除外しません。 これは、しかし、それはあなたにすべての既知のスプライス変異体を包含するプライマー対を与えることを意味するものではありません! あなたは単にあなたの目標基準を緩めています。
シーケンスを入力し、必要に応じてカスタマイズしたら、ページの一番下までスクロールし、”新しいグラフィックビューを使用”をチェックした後、”プライマーを これにより、提案されたプライマー対がターゲットを増幅する場所のマップと、プライマーの長さ、正確な位置、それぞれのTm、GC%、および自己相補性を反映するスコア(0.00は予測された相補性を反映していない)の分析が返されます。
ヒントThree:プライマーターゲットを予測する方法
あなたのプライマーがターゲット以外のものに当たったかどうかをどのように確認できますか? Primer-BLASTに移動します。 クエリボックスに、前方プライマー(5’から3’)を入力します。 次に、20Nを連続して入力して、プライマーを個々の重複していない整列に分離します。 Nの後、以下に示すように、リバースプライマー(5’から3’)を入力します。
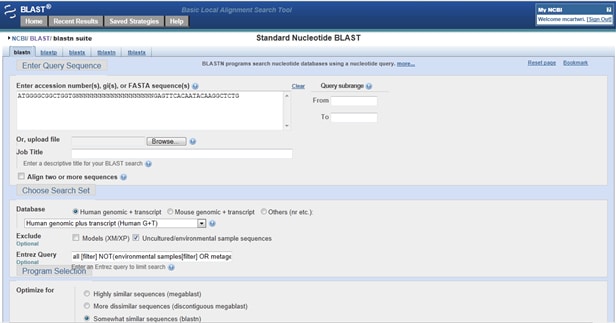
あなたの結果を得たら、ある組合せがあるようにそれらを点検しなさい。 フォワードプライマーがフォワードストランド(注釈付きストランドプラス/プラス)に整列し、リバースプライマーが同じヒットに整列しているが、リバースストランド(ストランドプラス/マイナス)に整列している場合、プライマーはそのヒットを増幅する可能性があります。
トリック二:あなたの結果は、オリーブヒヒやネアンデルタール人など、あなたのPCRサンプルを汚染している可能性は低いものが含まれていますか? 人間やマウスのサンプルを使用している場合は、データベースの下に指定されていることを確認してくださ または、特定の種を除外することもできます。
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
これはあなたを助けましたか? その後、あなたのネットワークと共有してくださ