L’algoritmo di base Local Alignment Search Tool (BLAST) è al centro di una suite gratuita di risorse online disponibili attraverso il National Center for Biotechnology Information (NCBI). Mentre la maggior parte dei ricercatori sono consapevoli di BLAST come strumento di allineamento sequenza, BLAST suite di NCBI offre molto di più! Illustrerò in modo approfondito come utilizzare queste risorse per individuare polimorfismi a singolo nucleotide (SNP) in un gene; progettare primer con Primer-BLAST; e convalidare gli obiettivi di primer.
Suggerimento uno: Come trovare SNPs
Data l’importanza degli SNPs sia nella malattia che nella ricerca, NCBI fornisce strumenti per la raccolta degli SNPS segnalati da un gene. Per trovare SNP, inizia dalla home page di NCBI e digitare il gene di interesse nella barra di ricerca. Seleziona SNP dal menu a discesa Tutti i database a sinistra della barra di ricerca, come mostrato di seguito:
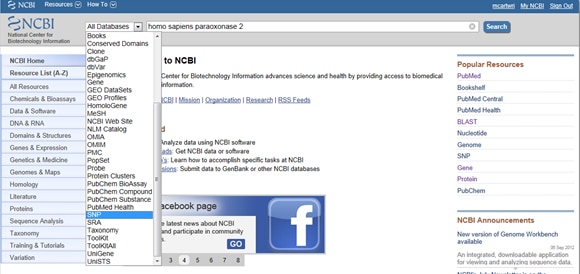
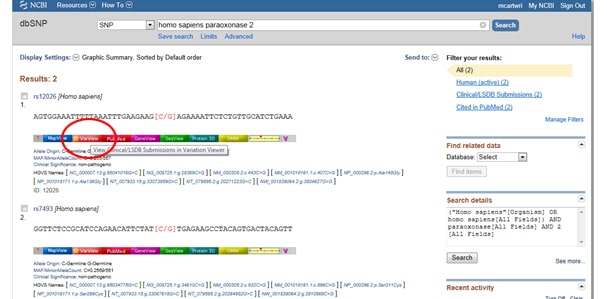
Trick One: hai bisogno di filtrare i risultati in modo che tu stia solo guardando i risultati clinicamente associati? Vai a quella casella di visualizzazione etichettata Filtri a destra della lista SNP sotto Variazione osservata. Dopo aver selezionato l’opzione di filtro, assicurati di premere il pulsante Aggiorna.
Suggerimento due: Come progettare Primer
NCBI fornisce Primer-BLAST per la progettazione automatica di primer in base a una sequenza di query. Per iniziare a progettare primer, vai alla homepage BLAST e scorri verso il basso fino all’opzione Primer-BLAST in Specialized BLAST. Inserisci la sequenza di destinazione per taglia e incolla o, se è elencata nei database NCBI, come numero di adesione. Copro alcune opzioni di personalizzazione di seguito, ma a questo punto, è possibile generare primer senza fare alcuna personalizzazione aggiuntiva!
Range: A destra della casella per l’inserimento della sequenza, è possibile specificare l’intervallo esatto (numerato da 5 ‘a 3’, dall’inizio della sequenza) del target che verrà considerato per la progettazione dei primer forward e reverse.
Usa il mio primer forward (5 ‘- > 3’ su più strand): Seleziona questo se hai già progettato i tuoi primer e vuoi che Primer-BLAST fornisca alcune analisi (ad esempio Tm) su di loro.
PCR formato del prodotto: Impostare l’intervallo di lunghezze accettabili dei prodotti PCR qui.
# di primer da restituire: imposta il numero preferito di set di primer candidati da considerare. Si noti che non è una garanzia, soprattutto se i parametri sono troppo severi o privi di senso (ad esempio, è stato specificato un prodotto sotto la dimensione del prodotto PCR che non può essere superiore a 500 bp, ma sotto l’intervallo si desidera considerare solo i primer più di 1 kb di distanza).
Temperature di fusione del primer: Ciò consente di specificare il Tm (per un rapido aggiornamento sulla temperatura di fusione, controllare i nostri suggerimenti per qPCR e regolare PCR primer design).
Intervallo di giunzione esone: se si desidera escludere il DNA genomico (dove gli esoni sono divisi per introni non codificanti), impostare questo su Primer deve estendersi su una giunzione esone-esone.
Controllo di specificità: a meno che non si desideri che Primer-BLAST restituisca primer che andranno fuori bersaglio (generalmente non raccomandato!), lascia questo controllato e specifica l’organismo da cui provengono i tuoi campioni e quale Database utilizzare, a seconda che tu stia prendendo di mira mRNA, gDNA, ecc. Abilitando il controllo della specificità, Primer-BLAST escluderà i primer che potrebbero amplificare qualcosa al di fuori della sequenza di destinazione.
Gestione delle varianti di giunzione: se si seleziona questa opzione, fattibile solo se si sta lavorando su sequenze di mRNA, Primer – BLAST non escluderà coppie di primer che potrebbero amplificare più varianti di giunzione mRNA del bersaglio. Questo non significa, tuttavia, che vi darà coppie di primer che comprendono tutte le varianti di giunzione noti! Stai semplicemente allentando i tuoi criteri di destinazione.
Una volta inserita la sequenza e personalizzata secondo necessità, scorrere verso il basso fino alla fine della pagina e, dopo aver controllato Usa nuova vista grafica, premere Get Primer. Questo restituirà una mappa di dove le coppie di primer suggerite amplificheranno il tuo target, così come le analisi sui primer: la loro lunghezza, posizione precisa, rispettiva Tm, GC% e punteggi che riflettono l’auto-complementarità (con 0.00 che non riflette alcuna complementazione prevista).
Suggerimento tre: Come prevedere gli obiettivi di primer
Come puoi controllare se i tuoi primer colpiscono qualcosa fuori bersaglio? Vai a Primer-BLAST. Nella casella query, inserire il primer (da 5’ a 3’). Ora digitare 20 N di fila per separare i primer in singoli allineamenti non sovrapposti. Dopo le N, inserisci il tuo primer inverso (anche da 5’ a 3’), come mostrato di seguito:
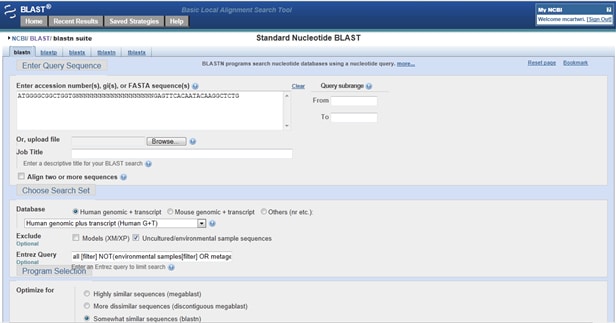
Una volta ottenuti i risultati, controllali per determinate combinazioni. Se il primer in avanti si allinea sul filo in avanti (Filo annotato più / Più) e il primer inverso si allinea allo stesso colpo, ma sul filo inverso (Filo più/Meno), allora i primer possono amplificare quel colpo.
Trucco due: I tuoi risultati includono cose che difficilmente hanno contaminato i tuoi campioni di PCR, come i babbuini d’oliva e i Neanderthal? Se stai lavorando con campioni umani o di mouse, assicurati di avere quelli specificati in Database. In alternativa, è possibile escludere specie specifiche.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
Questo ti ha aiutato? Quindi si prega di condividere con la rete.