L’algorithme BLAST (Basic Local Alignment Search Tool) est au cœur d’une suite gratuite de ressources en ligne disponibles via le Centre National d’Information sur les biotechnologies (NCBI). Alors que la plupart des chercheurs connaissent BLAST comme un outil d’alignement de séquences, la suite BLAST de NCBI offre bien plus! Je vais expliquer en profondeur comment utiliser ces ressources pour localiser les polymorphismes nucléotidiques simples (SNP) dans un gène; concevoir des amorces avec Amorce-EXPLOSION; et valider des cibles d’amorces.
Pointe Un: Comment trouver des SNP
Compte tenu de l’importance des SNP dans la maladie et la recherche, le NCBI fournit des outils pour rassembler les SNP rapportés d’un gène. Pour trouver des SNP, commencez sur la page d’accueil de NCBI et tapez votre gène d’intérêt dans la barre de recherche. Sélectionnez SNP dans le menu déroulant Toutes les bases de données à gauche de la barre de recherche, comme indiqué ci-dessous:
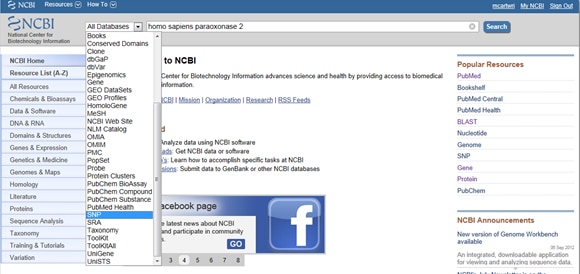
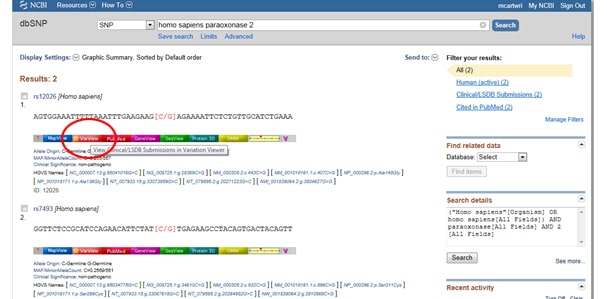
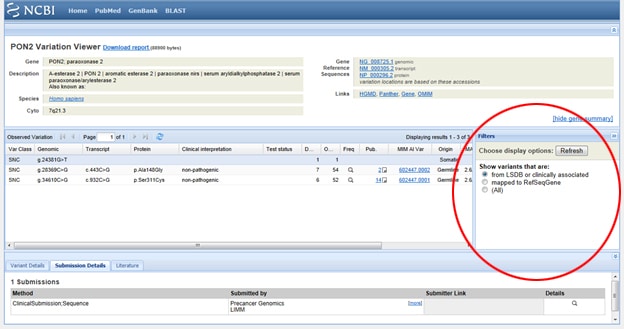
Première astuce: Vous devez filtrer les résultats afin de ne regarder que les résultats cliniquement associés? Accédez à cette boîte d’affichage intitulée Filtres à droite de la liste SNP sous Variation observée. Après avoir sélectionné votre option de filtre, assurez-vous d’appuyer sur le bouton Actualiser.
Astuce Deux: Comment concevoir des amorces
NCBI fournit une amorce-EXPLOSION pour concevoir automatiquement des amorces en fonction d’une séquence de requêtes. Pour commencer à concevoir des amorces, accédez à la page d’accueil de BLAST et faites défiler jusqu’à l’option Amorce -BLAST sous BLAST spécialisé. Entrez votre séquence cible soit par copier-coller, soit, si elle est répertoriée dans les bases de données de NCBI, en tant que numéro d’accession. Je couvre quelques options de personnalisation ci-dessous, mais à ce stade, vous pouvez générer des amorces sans faire de personnalisation supplémentaire!
Plage: À droite de la case de saisie de votre séquence, vous pouvez spécifier la plage exacte (numérotée de 5’à 3′, à partir du début de votre séquence) de la cible qui sera prise en compte pour la conception des amorces avant et arrière.
Utilisez mon propre primaire avant (5’- >3’sur le brin plus): Sélectionnez ceci si vous avez déjà conçu vos amorces et souhaitez que Primer-BLAST fournisse des analyses (par exemple Tm) à leur sujet.
Taille du produit PCR: Définissez ici la plage de longueurs acceptables des produits PCR.
# d’amorces à renvoyer: Cela définit votre nombre préféré d’ensembles d’amorces candidats à considérer. Notez que ce n’est pas une garantie, surtout si vos paramètres sont trop stricts ou absurdes (par exemple, vous avez spécifié un produit sous la taille du produit PCR qui ne peut pas dépasser 500 pb, mais sous la plage, vous ne voulez considérer que les amorces distantes de plus de 1 ko).
Températures de fusion de l’amorce: Cela vous permet de spécifier votre Tm (pour un rappel rapide sur la température de fusion, consultez nos conseils pour la conception d’amorces qPCR et PCR régulière).
Étendue de jonction d’exons: Si vous souhaitez exclure l’ADN génomique (où les exons sont divisés par des introns non codants), définissez-la sur Primer must span une jonction exon-exon.
Vérification de la spécificité: À moins que vous ne souhaitiez que Primer-BLAST renvoie des amorces qui seront hors cible (généralement déconseillées!), laissez cela vérifié et spécifiez l’organisme d’où proviennent vos échantillons ainsi que la base de données à utiliser, selon que vous ciblez l’ARNm, l’ADNGD, etc. En activant la vérification de la spécificité, Primer-BLAST exclura les amorces qui pourraient amplifier quelque chose en dehors de votre séquence cible.
Gestion des variantes d’épissure: Si vous sélectionnez cette option – uniquement réalisable si vous travaillez à partir de séquences d’ARNm – Primer-BLAST n’exclura pas les paires d’amorces qui pourraient amplifier plusieurs variantes d’épissure d’ARNm de votre cible. Cela ne signifie pas, cependant, qu’il vous donnera des paires d’amorces qui englobent toutes les variantes d’épissure connues! Vous relâchez simplement vos critères cibles.
Une fois que vous avez entré votre séquence et que vous l’avez personnalisée au besoin, faites défiler vers le bas de la page et, après avoir coché Utiliser une nouvelle vue graphique, appuyez sur Obtenir des amorces. Cela renverra une carte de l’endroit où les paires d’amorces suggérées amplifieront votre cible, ainsi que des analyses sur les amorces: leur longueur, leur emplacement précis, les Tm respectives, le% GC et les scores reflétant l’auto-complémentarité (avec 0,00 ne reflétant aucune complémentation prévue).
Troisième astuce: Comment prédire les cibles d’amorçage
Comment pouvez-vous vérifier si vos amorces atteignent quelque chose hors cible? Allez à Amorce-EXPLOSION. Dans la zone de requête, entrez votre amorce avant (5′ à 3’). Tapez maintenant 20 N d’affilée pour séparer les amorces en alignements individuels sans chevauchement. Après les N, entrez votre amorce inverse (également de 5’à 3’), comme indiqué ci-dessous:
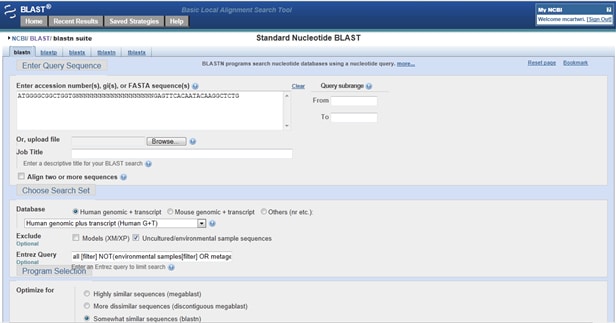
Une fois que vous avez obtenu vos résultats, vérifiez-les pour certaines combinaisons. Si votre amorce avant s’aligne sur le brin avant (brin annoté Plus / Plus) et que votre amorce inverse s’aligne sur le même coup, mais sur le brin inverse (Brin Plus / Moins), alors vos amorces peuvent amplifier ce coup.
Deuxième astuce: Vos résultats incluent-ils des éléments peu susceptibles d’avoir contaminé vos échantillons de PCR, tels que les babouins olive et les Néandertaliens? Si vous travaillez avec des échantillons humains ou de souris, assurez-vous d’avoir ceux spécifiés sous Base de données. Vous pouvez également exclure des espèces spécifiques.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
Cela vous a-t-il aidé? Ensuite, veuillez partager avec votre réseau.