El algoritmo de la Herramienta Básica de Búsqueda de Alineación Local (BLAST) está en el corazón de un conjunto gratuito de recursos en línea disponibles a través del Centro Nacional de Información Biotecnológica (NCBI). Si bien la mayoría de los investigadores son conscientes de que BLAST es una herramienta de alineación de secuencias, ¡la suite BLAST de NCBI ofrece mucho más! Cubriré en profundidad cómo usar estos recursos para localizar polimorfismos de un solo nucleótido (SNP) en un gen; diseñar cebadores con explosión de cebador; y validar objetivos de cebador.
Punta Uno: Cómo encontrar SNPs
Dada la importancia de los SNPs tanto en la enfermedad como en la investigación, el NCBI proporciona herramientas para cotejar los SNPs reportados de un gen. Para encontrar SNPs, comience en la página de inicio de NCBI y escriba su gen de interés en la barra de búsqueda. Seleccione SNP en el menú desplegable Todas las bases de datos a la izquierda de la barra de búsqueda, como se muestra a continuación:
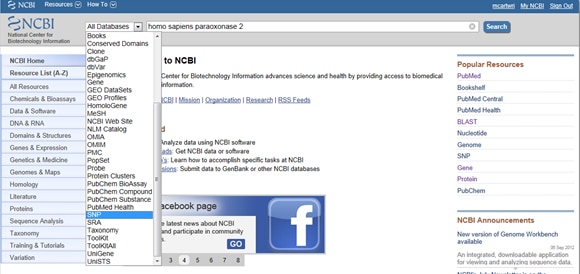
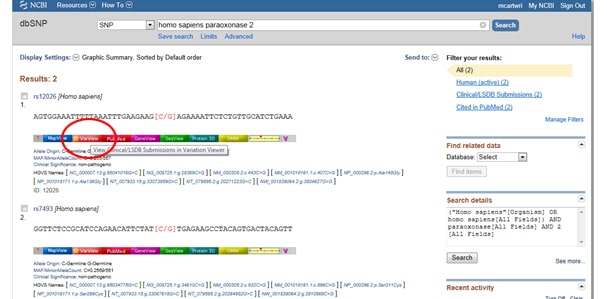
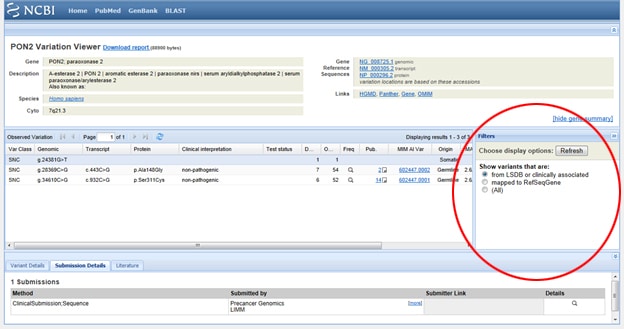
Truco Uno: ¿Necesitas filtrar los resultados para que solo veas resultados clínicamente asociados? Vaya a ese cuadro de visualización etiquetado Filtros a la derecha de la lista SNP en Variación observada. Después de seleccionar la opción de filtro, asegúrate de pulsar el botón Actualizar.
Consejo Dos: Cómo diseñar Imprimaciones
NCBI proporciona un Primer-BLAST para diseñar automáticamente imprimaciones basadas en una secuencia de consulta. Para empezar a diseñar imprimaciones, ve a la página de inicio de la EXPLOSIÓN y desplázate hacia abajo hasta la opción Imprimación-EXPLOSIÓN en EXPLOSIÓN especializada. Ingrese su secuencia de destino, ya sea cortando y pegando o, si aparece en las bases de datos de NCBI, como un número de acceso. Cubro algunas opciones de personalización a continuación, pero en este punto, puede generar cebadores sin hacer ninguna personalización adicional.
Rango: A la derecha de la casilla para ingresar su secuencia, puede especificar el rango exacto (numerado de 5′ a 3′, desde el inicio de su secuencia) del objetivo que se considerará para diseñar los cebadores hacia adelante y hacia atrás.
Use mi propia imprimación directa (5′->3′ en la cadena plus): Seleccione esto si ya ha diseñado sus imprimaciones y desea que Primer-BLAST proporcione algunos análisis (por ejemplo, Tm) sobre ellas.
Tamaño del producto de PCR: Establezca el rango de longitudes aceptables de los productos de PCR aquí.
# de cebadores a devolver: Esto establece el número preferido de conjuntos de cebadores candidatos a considerar. Tenga en cuenta que no es una garantía, especialmente si sus parámetros son demasiado estrictos o sin sentido (por ejemplo, especificó un producto bajo tamaño de producto PCR que no puede ser superior a 500 pb, pero bajo rango, solo desea considerar cebadores con más de 1 kb de distancia).
Temperaturas de fusión de imprimación: Esto le permite especificar su Tm (para un rápido repaso de la temperatura de fusión, consulte nuestros consejos para el diseño de imprimación qPCR y PCR regular).
Intervalo de unión de exones: Si desea excluir el ADN genómico (donde los exones se dividen por intrones no codificantes), establezca esto en Primer debe abarcar una unión exón-exón.
Comprobación de especificidad: A menos que desee que Cebador-BLAST devuelva cebadores que se desviarán del objetivo (¡generalmente no se recomienda!), deje esto marcado y especifique el organismo del que provienen sus muestras, así como qué Base de datos usar, dependiendo de si está apuntando a mRNA, gDNA, etc. Al habilitar la comprobación de especificidad, la explosión de cebador excluirá los cebadores que podrían amplificar algo fuera de la secuencia de destino.
Manejo de variantes de empalme: Si selecciona esta opción, solo factible si está trabajando con secuencias de ARNm, entonces Imprimación – EXPLOSIÓN no excluirá los pares de cebadores que podrían amplificar múltiples variantes de empalme de ARNm de su objetivo. Esto no significa, sin embargo, que le dará pares de imprimación que abarcan todas las variantes de empalme conocidas. Simplemente estás aflojando los criterios de tu objetivo.
Una vez que hayas introducido la secuencia y la hayas personalizado según sea necesario, desplázate hacia abajo hasta la parte inferior de la página y, después de comprobar Usar nueva vista gráfica, pulsa Obtener Cebadores. Esto devolverá un mapa de dónde los pares de cebadores sugeridos amplificarán su objetivo, así como análisis de los cebadores: su longitud, ubicación precisa, Tm respectivas, GC% y puntuaciones que reflejan auto complementariedad (con 0.00 que refleja ninguna complementación prevista).
Consejo tres: Cómo predecir Objetivos de Imprimación
¿Cómo puedes comprobar si tus cebadores alcanzan algo fuera de objetivo? Ve a EXPLOSIÓN de Cebador. En el cuadro de consulta, ingrese su primer delantero (5′ a 3′). Ahora escriba 20 N seguidas para separar los cebadores en alineaciones individuales que no se superpongan. Después de las N, ingrese su imprimación inversa (también de 5′ a 3′), como se muestra a continuación:
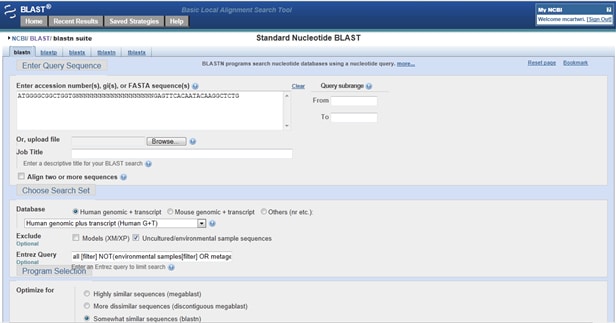
Una vez que obtenga sus resultados, compruebe si hay ciertas combinaciones. Si el cebador de avance se alinea en la hebra hacia adelante (anotado Strand Plus/Plus) y su primer revés se alinea con el mismo éxito, pero en el reverso strand (Capítulo Más/Menos), entonces su cebadores pueden amplificar ese golpe.
Truco Dos: ¿Sus resultados incluyen cosas que probablemente no hayan contaminado sus muestras de PCR, como babuinos de oliva y neandertales? Si está trabajando con muestras de humanos o ratones, asegúrese de tener las especificadas en Base de datos. Alternativamente, puede excluir especies específicas.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
Tiene esto ha ayudado? Entonces, por favor, comparta con su red.