Der grundlegende BLAST-Algorithmus (Local Alignment Search Tool) ist das Herzstück einer kostenlosen Suite von Online-Ressourcen, die über das National Center for Biotechnology Information (NCBI) verfügbar sind. Während die meisten Forscher BLAST als Sequenzausrichtungswerkzeug kennen, bietet die BLAST Suite von NCBI so viel mehr! Ich werde ausführlich behandeln, wie diese Ressourcen verwendet werden, um Einzelnukleotidpolymorphismen (SNPs) in einem Gen zu lokalisieren; Primer mit Primer-BLAST entwerfen; und Primer-Targets validieren.
Tipp Eins: So finden Sie SNPs
Angesichts der Bedeutung von SNPs sowohl für Krankheiten als auch für die Forschung bietet NCBI Tools zum Zusammenstellen der gemeldeten SNPs eines Gens. Um SNPs zu finden, beginnen Sie auf der NCBI-Homepage und geben Sie Ihr Interessengebiet in die Suchleiste ein. Wählen Sie SNP aus dem Dropdown-Menü Alle Datenbanken links neben der Suchleiste aus, wie unten gezeigt:
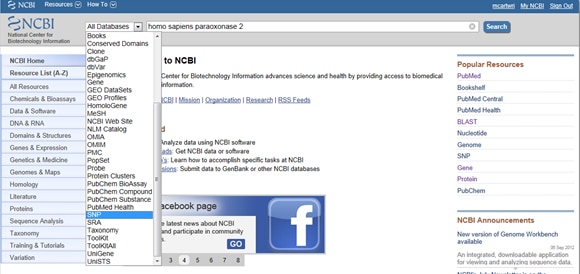
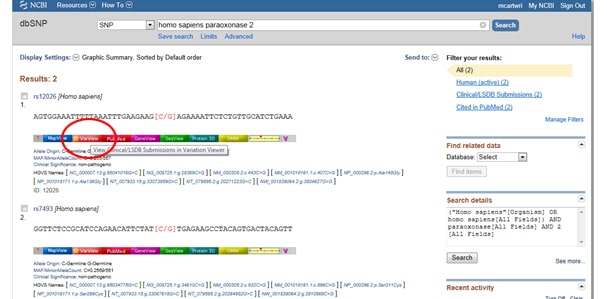
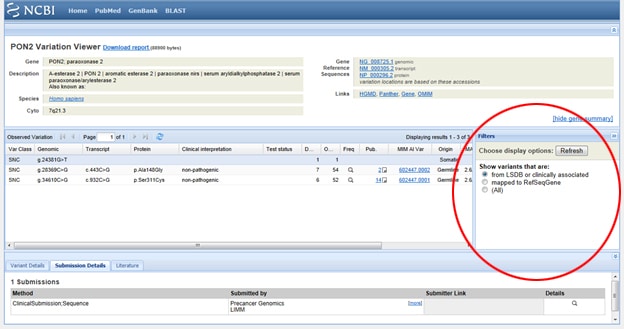
Trick eins: Müssen Sie die Ergebnisse so filtern, dass Sie nur klinisch assoziierte Ergebnisse betrachten? Gehen Sie zu diesem Anzeigefeld mit der Bezeichnung Filter rechts neben der SNP-Liste unter Beobachtete Variation. Nachdem Sie Ihre Filteroption ausgewählt haben, klicken Sie auf die Schaltfläche Aktualisieren.
Tipp zwei: Primer entwerfen
NCBI bietet Primer-BLAST zum automatischen Entwerfen von Primern basierend auf einer Abfragesequenz. Um mit dem Entwerfen von Primern zu beginnen, gehen Sie zur BLAST-Homepage und scrollen Sie unter Specialized BLAST zur Option Primer-BLAST. Geben Sie Ihre Zielsequenz entweder durch Ausschneiden und Einfügen oder, wenn sie in den Datenbanken der NCBI aufgeführt ist, als Beitrittsnummer ein. Ich behandle einige Anpassungsoptionen unten, aber an dieser Stelle können Sie Primer generieren, ohne zusätzliche Anpassungen vorzunehmen!
Bereich: Rechts neben dem Feld für die Eingabe Ihrer Sequenz können Sie den genauen Bereich (nummeriert 5′ bis 3′, vom Beginn Ihrer Sequenz an) des Ziels angeben, das für den Entwurf der Vorwärts- und Rückwärtsprimer berücksichtigt wird.
Verwenden Sie meinen eigenen Vorwärtsprimer (5′->3′ auf plus Strang): Wählen Sie diese Option, wenn Sie Ihre Primer bereits entworfen haben und möchten, dass Primer-BLAST einige Analysen (z. B. Tm) über sie bereitstellt.
PCR-Produktgröße: Stellen Sie hier den Bereich der zulässigen Längen der PCR-Produkte ein.
Anzahl der zurückzugebenden Primer: Hiermit wird die bevorzugte Anzahl der zu berücksichtigenden Kandidatensätze von Primern festgelegt. Beachten Sie, dass dies keine Garantie ist, insbesondere wenn Ihre Parameter zu streng oder unsinnig sind (z. B. haben Sie ein Produkt unter PCR-Produktgröße angegeben, das nicht mehr als 500 bp betragen darf, aber unter Bereich möchten Sie nur Primer mit einem Abstand von mehr als 1 kb berücksichtigen).
Primer-Schmelztemperaturen: Auf diese Weise können Sie Ihre Tm angeben (für eine schnelle Auffrischung der Schmelztemperatur lesen Sie unsere Tipps für qPCR und reguläres PCR-Primer-Design).
Exon junction span: Wenn Sie genomische DNA ausschließen möchten (wobei Exons durch nicht kodierende Introns geteilt werden), setzen Sie dies auf Primer muss einen Exon-Exon-Übergang überspannen.
Spezifitätsprüfung: Es sei denn, Sie möchten, dass Primer-BLAST Primer zurückgibt, die vom Ziel abweichen (im Allgemeinen nicht empfohlen!), lassen Sie diese Option aktiviert und geben Sie den Organismus an, aus dem Ihre Proben stammen, sowie die zu verwendende Datenbank, je nachdem, ob Sie auf mRNA, gDNA usw. abzielen. Durch Aktivieren der Spezifitätsprüfung schließt Primer-BLAST Primer aus, die etwas außerhalb Ihrer Zielsequenz amplifizieren könnten.
Handhabung von Spleißvarianten: Wenn Sie diese Option auswählen – nur möglich, wenn Sie mit mRNA–Sequenzen arbeiten -, schließt Primer-BLAST Primerpaare nicht aus, die mehrere mRNA-Spleißvarianten Ihres Ziels amplifizieren könnten. Dies bedeutet jedoch nicht, dass Sie Primerpaare erhalten, die alle bekannten Spleißvarianten umfassen! Sie lockern einfach Ihre Zielkriterien.
Sobald Sie Ihre Sequenz eingegeben und nach Bedarf angepasst haben, scrollen Sie zum Ende der Seite und klicken Sie nach dem Aktivieren von Neue grafische Ansicht verwenden auf Primer abrufen. Dies gibt eine Karte zurück, wo die vorgeschlagenen Primerpaare Ihr Ziel verstärken, sowie Analysen zu den Primern: ihre Länge, genaue Position, jeweilige Tms, GC% und Werte, die die Selbstkomplementarität widerspiegeln (wobei 0,00 keine vorhergesagte Komplementarität widerspiegelt).
Tipp drei: Primerziele vorhersagen
Wie können Sie überprüfen, ob Ihre Primer etwas außerhalb des Ziels treffen? Gehen Sie zu Primer-BLAST. Geben Sie im Abfragefeld Ihren Forward Primer (5′ bis 3′) ein. Geben Sie nun 20 N’s hintereinander ein, um die Primer in einzelne, nicht überlappende Alignments zu trennen. Geben Sie nach den N’s Ihren umgekehrten Primer (ebenfalls 5 ‚bis 3‘) ein, wie unten gezeigt:
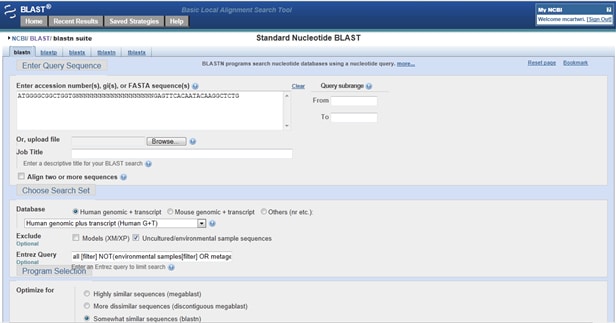
Sobald Sie Ihre Ergebnisse erhalten haben, überprüfen Sie sie auf bestimmte Kombinationen. Wenn Ihr Vorwärtsprimer am Vorwärtsstrang ausgerichtet ist (kommentierter Strang Plus / Plus) und Ihr Rückwärtsprimer am selben Treffer ausgerichtet ist, jedoch am Rückwärtsstrang (Strang Plus / Minus), können Ihre Primer diesen Treffer verstärken.Trick zwei: Enthalten Ihre Ergebnisse Dinge, die Ihre PCR-Proben wahrscheinlich nicht kontaminiert haben, wie Olivenpaviane und Neandertaler? Wenn Sie mit Proben von Menschen oder Mäusen arbeiten, stellen Sie sicher, dass diese unter Datenbank angegeben sind. Alternativ können Sie bestimmte Arten ausschließen.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
Hat Ihnen das geholfen? Dann teilen Sie bitte mit Ihrem Netzwerk.