den grundläggande local Alignment Search Tool (BLAST) algoritm är kärnan i en gratis svit av online-resurser tillgängliga via National Center for Biotechnology Information (NCBI). Medan de flesta forskare är medvetna om BLAST som ett sekvensinriktningsverktyg, erbjuder NCBIS BLAST suite så mycket mer! Jag kommer att täcka djupt hur man använder dessa resurser för att lokalisera enstaka nukleotidpolymorfismer (SNP) i en gen; designa primers med Primer-BLAST; och validera primermål.
tips ett: Hur man hittar SNPs
Med tanke på vikten av SNPs i både sjukdom och forskning, tillhandahåller NCBI verktyg för att sammanställa en gens rapporterade SNPs. För att hitta SNP: er, börja på NCBIS hemsida och skriv din gen av intresse i sökfältet. Välj SNP från rullgardinsmenyn Alla databaser till vänster om sökfältet, som visas nedan:
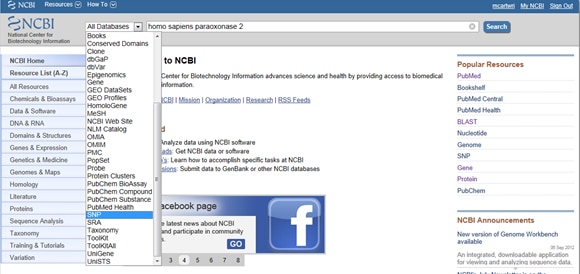
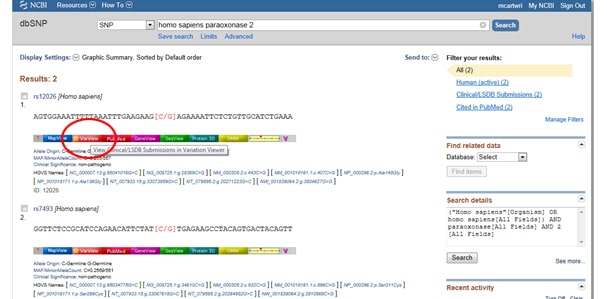
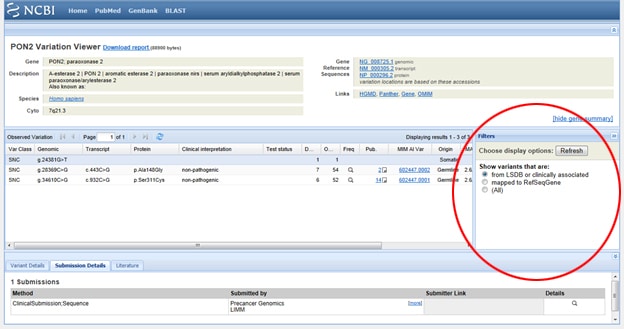
Trick One: behöver du filtrera resultaten så att du bara tittar på kliniskt associerade resultat? Gå till den visningsrutan märkta filter till höger om SNP-listan under observerad Variation. När du har valt ditt filteralternativ, se till att trycka på uppdateringsknappen.
Tips två: Hur man utformar Primers
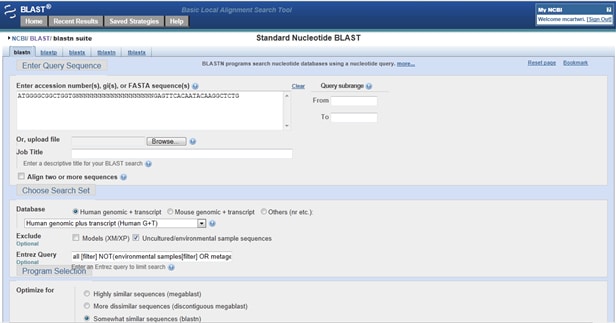
NCBI ger Primer-BLAST för att automatiskt utforma primers baserat på en frågesekvens. För att börja designa primers, gå till BLAST hemsida och bläddra ner till Primer-BLAST alternativet under specialiserad BLAST. Ange din målsekvens antingen genom cut-and-paste eller, om den är listad i NCBI: s databaser, som ett anslutningsnummer. Jag täcker några anpassningsalternativ nedan, men vid denna tidpunkt kan du generera primers utan att göra någon ytterligare anpassning!
intervall: till höger om rutan för att ange din sekvens kan du ange det exakta intervallet (som numrerat 5′ till 3′, från början av din sekvens) för målet som kommer att övervägas för att utforma framåt-och bakåtprimrarna.
använd min egen Forward primer (5′->3′ på plus strand): Välj detta om du redan har designat dina primers och vill att Primer-BLAST ska ge några analyser (t.ex. Tm) om dem.
PCR Produktstorlek: Ställ in intervallet för acceptabla längder för PCR-produkterna här.
# of primers to return: detta anger ditt önskade antal kandidatuppsättningar primers att överväga. Observera att det inte är en garanti, särskilt om dina parametrar är för strikta eller meningslösa (t.ex. du angav en produkt under PCR-Produktstorlek som inte kan vara mer än 500 bp, men under intervallet vill du bara överväga primers mer än 1 kb från varandra).
Primersmältningstemperaturer: Detta låter dig ange din Tm (för en snabb uppdatering av smälttemperaturen, kolla in våra tips för qPCR och vanlig PCR-primerdesign).
Exon junction span: om du vill utesluta genomiskt DNA (där exoner delas av icke-kodande introner), ställ sedan in detta till Primer måste spänna över en exon-exon junction.
Specificitetskontroll: om du inte vill att Primer-BLAST ska returnera primers som kommer att gå utanför målet (rekommenderas vanligtvis inte!), lämna detta kontrollerat och ange organismen som dina prover kommer från samt vilken databas som ska användas, beroende på om du riktar dig mot mRNA, gDNA, etc. Genom att aktivera specificitetskontrollen kommer Primer-BLAST att utesluta primers som kan förstärka något utanför din målsekvens.
Splice variant hantering: om du väljer det här alternativet – endast möjligt om du arbetar av mRNA – sekvenser-kommer Primer-BLAST inte att utesluta primerpar som kan förstärka flera mRNA splice-varianter av ditt mål. Detta betyder dock inte att det kommer att ge dig primerpar som omfattar alla kända skarvvarianter! Du löser helt enkelt dina målkriterier.
När du har angett din sekvens och anpassat efter behov, bläddra ner till botten av sidan och, efter att ha kontrollerat Använd ny grafisk vy, tryck på få Primers. Detta kommer att returnera en karta över var de föreslagna primerparen förstärker ditt mål, liksom analyser på primrarna: deras längd, exakta plats, respektive Tm, GC% och poäng som återspeglar självkomplementaritet (med 0,00 som återspeglar ingen förutsagd komplementering).
Tips Tre: hur man förutspår Primermål
hur kan du kontrollera om dina primers träffar något utanför målet? Gå till Primer-BLAST. I rutan fråga anger du din Forward primer (5′ till 3′). Skriv nu in 20 N i rad för att separera primrarna i individuella, icke-överlappande inriktningar. Efter N: erna anger du din omvända primer (även 5′ till 3′), som visas nedan:
När du får dina resultat, kontrollera dem för vissa kombinationer. Om din främre primer är inriktad på den främre strängen (annoterad sträng Plus/Plus) och din omvända primer är inriktad på samma träff, men på den omvända strängen (sträng Plus/Minus), kan dina primers förstärka den träffen.
Trick Two: innehåller dina resultat saker som sannolikt inte har förorenat dina PCR-prover, såsom olivbabianer och neandertalare? Om du arbetar med mänskliga eller musprover, se till att du har de som anges under databasen. Alternativt kan du utesluta specifika arter.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
har detta hjälpt dig? Vänligen dela med ditt nätverk.