기본적인 로컬 맞춤 검색 툴(폭)알고리즘을의 중심에서의 무료 스위트 온라인 사용할 수 있는 리소스를 통해 국내 센터를 위한 생명공학 정보(NCBI). 대부분의 연구자들이 시퀀스 정렬 도구로 BLAST 를 알고 있지만 NCBI 의 BLAST suite 는 훨씬 더 많은 것을 제공합니다! 내가 커버에서 사용하는 방법을 심도있게 이러한 자원을 찾을 단일 염기 polymorphisms(SNPs)에서는 유전자,디자인 프라이머는 프라이머-폭파;그리고 유효성 검사 프라이머 대상입니다.
팁 하나: Snp 를 찾는 방법
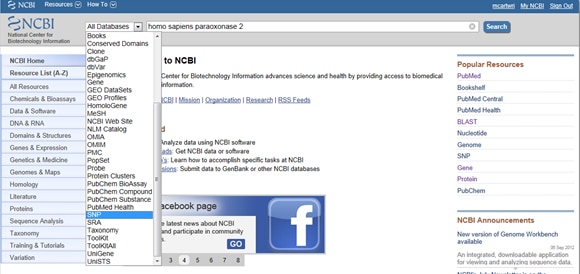
질병과 연구 모두에서 Snp 의 중요성을 감안할 때 NCBI 는 유전자의보고 된 Snp 를 대조하기위한 도구를 제공합니다. Snp 를 찾으려면 NCBI 의 홈페이지에서 시작하여 검색 창에 관심있는 유전자를 입력하십시오. 선택 SNP 에서 모든 데이터베이스 드롭다운 메뉴를 왼쪽의 검색 표시줄 아래와 같이
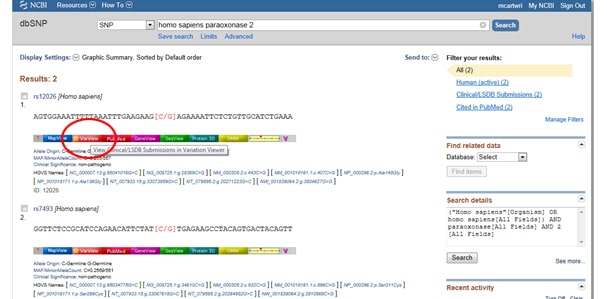
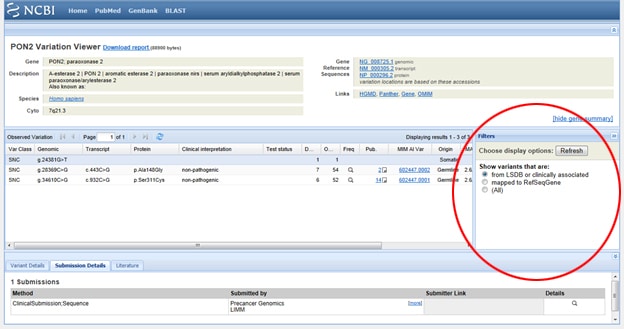
Trick One:임상 적으로 관련된 결과 만보고 있도록 결과를 필터링해야합니까? 관찰 된 변형 아래의 SNP 목록 오른쪽에있는 필터라고 표시된 표시 상자로 이동하십시오. 필터 옵션을 선택한 후에는 새로 고침 버튼을 눌러야합니다.
팁:는 방법을 디자인하는 프라이머
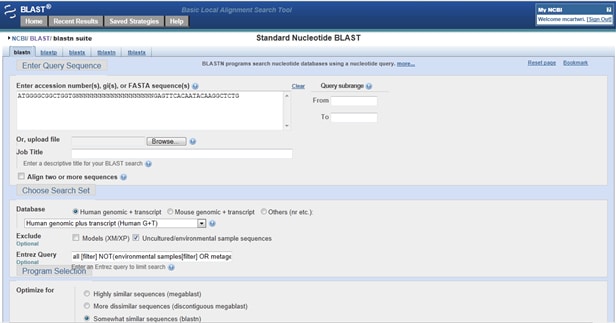
NCBI 에서 제공하는 프라이머는 폭발을 위해 자동으로 디자인 프라이머는 쿼리를 기반으로 순서에 있습니다. 프라이머 디자인을 시작하려면 BLAST 홈페이지로 이동하여 Specialized BLAST 아래의 Primer-BLAST 옵션으로 아래로 스크롤하십시오. 잘라 내기 및 붙여 넣기로 대상 시퀀스를 입력하거나 NCBI 의 데이터베이스에 나열된 경우 가입 번호로 입력하십시오. 아래의 몇 가지 사용자 정의 옵션을 다루지 만이 시점에서 추가 사용자 정의를하지 않고도 프라이머를 생성 할 수 있습니다!
범위:의 오른쪽에 있는 상자를 입력할 순서를 지정할 수 있습니다 정확한 범위(으로 번호 5’3’에서 시작 시퀀스)의 대상이 될 것으로 간주를 위한 설계방향 및 역방향 프라이머.
를 사용하여 내 자신의 앞으로 프라이머(5′->3 에’더 스트랜드):Select 이 경우 당신은 이미 설계 당신의 프라이머 및 프라이머는 폭발을 제공하는 몇 가지 분석(예를 들어 Tm)습니다.
PCR 제품 크기:PCR 제품의 허용 가능한 길이 범위를 여기에서 설정하십시오.반환 할 프라이머의
#:이것은 고려해야 할 프라이머의 후보 세트의 선호하는 수를 설정합니다. 하지 않습을 보장하는 경우에 특히 당신의 매개 변수는 너무 엄격한 또는 무의미한(예를 들어,당신은 지정된 제품에서 PCR 제품 크기할 수 없는 500 개 이상의 bp,하지만 범위에서 당신만을 원하는 고려하는 프라이머 1 개 이상의 kb 별도).
프라이머 용융 온도: 이렇게하면 Tm 을 지정할 수 있습니다(용융 온도에 대한 빠른 재교육을 위해 qPCR 및 일반 PCR 프라이머 디자인에 대한 팁을 확인하십시오).
Exon junction 범위:원하는 경우를 제외하 genomic DNA(는 exons 별로 나뉘어 있 non-코딩 introns)설정한 다음 이를 뇌관해야 범위인 exon-exon junction.
특이성 체크인:원하지 않는 경우 프라이머는 돌풍을 반환 프라이머는 것입니다 이동 목표(일반적으로 사용하지 않는 것이 좋습니다!),이 확인하고 지정 생물 샘플에서뿐만 아니라 데이터베이스를 사용하는 경우에 따라 타겟 mRNA,도록 설계되었,etc. 특이성 검사를 가능하게함으로써,프라이머-블라스트는 목표 시퀀스 외부의 무언가를 증폭시킬 수있는 프라이머를 배제합니다.
스플라이스 변종 처리:이 옵션을 선택 하는 경우에만 가능한 작업하는 경우의 mRNA 시퀀스–다 프라이머는 폭발 제외되지 않습니다 프라이머 쌍할 수 있는 증폭 여러 mRNA 스플라이스 변종의 대상입니다. 그러나 이것은 알려진 모든 스플 라이스 변형을 포괄하는 프라이머 쌍을 제공한다는 의미는 아닙니다! 당신은 단순히 목표 기준을 풀고 있습니다.
일단 당신이 입력한 시퀀스로 주문을 받아서 만들어 필요한,아래로 스크롤하는 페이지 맨 아래 그리고,확인한 후 사용하여 새로운 그래픽 보기,히트 프라이머를 얻을. 이를 반환하지도는 제안된 프라이머 쌍 증폭하고 대상뿐만 아니라,분석에서 프라이머:의 길이를,정확한 위치,각 Tm 의,GC%,과 점수를 반영하는 자가 상보성(와 0.00 을 반영하지 않 예측 보완성).
팁 3:프라이머 타겟을 예측하는 방법
프라이머가 오프 타겟에 닿았는지 어떻게 확인할 수 있습니까? 프라이머-블라스트로 이동하십시오. 쿼리 상자에 앞으로 입문서(5’~3′)를 입력하십시오. 이제 프라이머를 개별 겹치지 않는 정렬로 분리하기 위해 20N 을 연속으로 입력하십시오. 후 N,입력하신의 역 프라이머(또한 5’3′),다음과 같습니다:
일단 결과를 얻으면 특정 조합이 있는지 확인하십시오. 하는 경우 앞으로 프라이머를 정렬하기에 앞으로 스트랜드(주석 Strand Plus/플러스)과 역방향 프라이머를 정렬은 같은 치하지만,반대로 가닥(Strand 플러스/마이너스),그 다음 당신의 프라이머는 증폭 수 있습는다.
속 두:당신의 결과를 포함한 것을 가능성이 있다고 오염의 PCR 샘플 올리브와 같은 원숭하고 네안데르탈 인? 인간 또는 마우스 샘플로 작업하는 경우 데이터베이스 아래에 지정된 샘플이 있는지 확인하십시오. 또는 특정 종을 제외시킬 수도 있습니다.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
이러분에게 도움이 되었는가? 그런 다음 네트워크와 공유하십시오.