használatához az alapvető helyi igazítási kereső eszköz (BLAST) algoritmus a Nemzeti Biotechnológiai Információs Központon (NCBI) keresztül elérhető online erőforrások ingyenes csomagjának középpontjában áll. Míg a legtöbb kutató tudatában van a BLASTNAK, mint szekvencia-Igazító eszköznek, az NCBI BLAST suite sokkal többet kínál! Részletesen kitérek arra, hogyan használhatom ezeket az erőforrásokat egyetlen nukleotid polimorfizmus (SNP) megtalálására egy génben; Primer-blasztos primerek tervezése; és alapozó célok érvényesítése.
Tip One: Hogyan lehet megtalálni az SNP-ket
tekintettel az SNP-k fontosságára mind a betegségben, mind a kutatásban, az NCBI eszközöket biztosít a gén bejelentett SNP-jeinek összegyűjtéséhez. Az SNP-k megtalálásához kezdje el az NCBI honlapján, majd írja be az érdeklődő génjét a keresősávba. Válassza ki az SNP-t az összes adatbázis legördülő menüből a keresősáv bal oldalán, az alábbiak szerint:
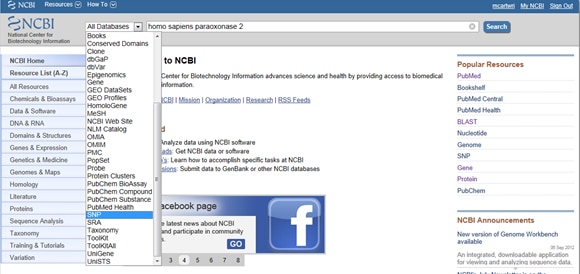
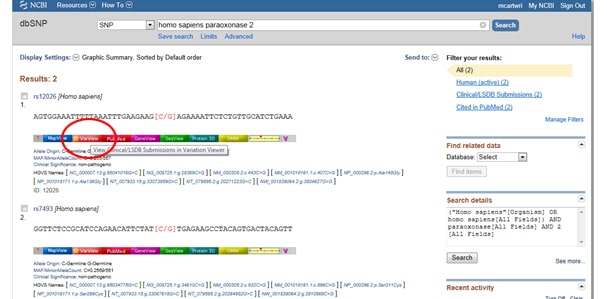
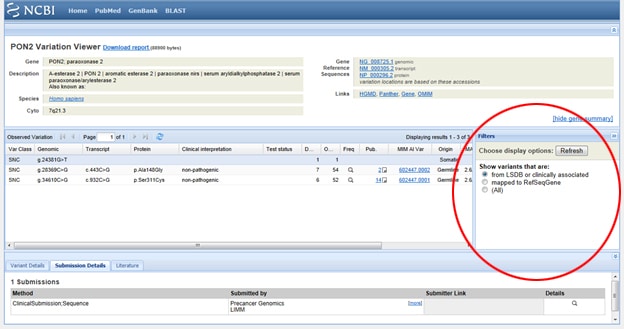
Trick One: szűrnie kell az eredményeket, hogy csak klinikailag kapcsolódó eredményeket nézzen? LÉPJEN az SNP lista jobb oldalán található szűrők feliratú megjelenítő dobozba a megfigyelt variáció alatt. Miután kiválasztotta a szűrő opciót, feltétlenül nyomja meg a Frissítés gombot.
tipp két: Hogyan tervezzünk primerek
NCBI biztosít Primer-BLAST automatikus tervezése primerek alapján egy lekérdezési sorrendben. A primerek tervezésének megkezdéséhez keresse fel a BLAST honlapját, majd görgessen le a Primer-BLAST opcióhoz a Specialized BLAST alatt. Adja meg a célsorozatot cut-and-paste vagy, ha szerepel az NCBI adatbázisaiban, csatlakozási számként. Az alábbiakban néhány testreszabási lehetőséget fedek le, de ezen a ponton alapozókat hozhat létre további testreszabás nélkül!
tartomány: a szekvencia megadásához szükséges doboz jobb oldalán megadhatja annak a célnak a pontos tartományát (5′ – től 3′ – ig számozva, a szekvencia kezdetétől), amelyet figyelembe kell venni az előre-hátra alapozók tervezéséhez.
használja a saját forward primert (5′ – >3 ‘ on plus strand): válassza ezt, ha már tervezte a primereket, és szeretné, hogy a Primer-BLAST néhány elemzést (pl. Tm) biztosítson róluk.
PCR Termékméret: itt állítsa be a PCR termékek elfogadható hosszúságainak tartományát.
# a primerek vissza: ez beállítja a kívánt számú jelölt készlet primers figyelembe venni. Vegye figyelembe, hogy ez nem garancia, különösen akkor, ha a paraméterei túl szigorúak vagy értelmetlenek (például megadott egy terméket a PCR Termékméret alatt, amely nem lehet több, mint 500 bp, de a tartomány alatt csak 1 kb-nál nagyobb alapozókat szeretne figyelembe venni).
Primer olvadási hőmérséklet: Ez lehetővé teszi, hogy adja meg a Tm (egy gyors frissítő olvadási hőmérséklet, nézd meg a tippeket qPCR rendszeres PCR primer kialakítás).
Exon junction span: ha ki akarja zárni a genomikus DNS-t (ahol az exonokat nem kódoló intronok osztják el), akkor állítsa ezt alapozónak kell lennie egy exon-exon csomópontra.
specificitás ellenőrzés: hacsak nem akarja, hogy a Primer-BLAST visszatérjen a célon kívüli alapozókhoz (általában nem ajánlott!), hagyja ezt ellenőrizni, és adja meg a szervezet a minták jönnek, valamint, hogy melyik adatbázist kell használni, attól függően, hogy ha célzás mRNA, gDNA, stb .. A specificitás ellenőrzésének engedélyezésével a Primer-BLAST kizárja azokat az alapozókat, amelyek erősíthetnek valamit a célsorozaton kívül.
Splice variant handling: ha ezt az opciót választja-csak akkor lehetséges, ha mRNS-szekvenciákkal dolgozik -, akkor a Primer-BLAST nem zárja ki azokat az alapozópárokat, amelyek felerősíthetik a cél több mRNA splice változatát. Ez azonban nem jelenti azt, hogy primer párokat fog adni, amelyek magukban foglalják az összes ismert splice változatot! Csak lazítasz a cél kritériumokon.
miután megadta a szekvenciát, és szükség szerint testre szabta, görgessen le az oldal aljára, majd az új grafikus nézet használatának ellenőrzése után nyomja meg a Get Primers gombot. Ez vissza fog térni egy térképet, ahol a javasolt primer párok felerősíti a cél, valamint az analytics az alapozók: a hossza, pontos helyszín, megfelelő Tm van, GC% – a, illetve pontszámok tükröző egyéni kiegészítő (a 0.00 tükröző nem előre complementation).
tipp három: hogyan lehet megjósolni Primer célok
hogyan lehet ellenőrizni, ha a primerek hit semmit off-cél? Menj a Primer-robbanás. A lekérdezés mezőbe írja be a forward primert (5′ to 3′). Most írja be a 20 N-t egymás után, hogy a primereket különálló, nem átfedő nyomvonalakra bontsa. Miután a N írja be a reverse primer (is 5′ 3′), az alábbiak szerint:
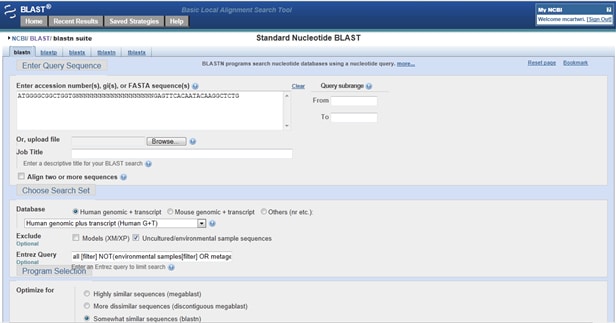
miután megkapta az eredményeket, ellenőrizze, hogy vannak-e bizonyos kombinációk. Ha a forward primer igazítja az előre strand (jegyzetekkel ellátott Strand Plus/Plus), a reverz primer igazítja, hogy ugyanaz a hit, de a fordított strand (Strand Plusz/Mínusz), akkor a primerek lehet erősíteni, hogy a hit.
Trick Two: az eredmények olyan dolgokat tartalmaznak, amelyek valószínűleg nem szennyezték a PCR-mintákat, például olívapáviánok és neandervölgyiek? Ha emberi vagy egérmintákkal dolgozik, győződjön meg róla, hogy rendelkezik az adatbázis alatt megadottakkal. Alternatív megoldásként kizárhat bizonyos fajokat.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
Ez segített Önnek? Akkor kérjük, ossza meg a hálózat.