paikallisen linjauksen Perusalgoritmin (BLAST) ytimessä on ilmainen online-resurssisarja, joka on saatavilla National Center for Biotechnology Informationin (NCBI) kautta. Vaikka useimmat tutkijat ovat tietoisia BLAST kuin sekvenssin kohdistus työkalu, NCBI: N BLAST suite tarjoaa niin paljon enemmän! Tutkin perusteellisesti, miten näitä resursseja voidaan käyttää yksittäisten nukleotidipolymorfismien (SNP) paikantamiseen geenissä; suunnittelen primerit Primer-Blastilla; ja validoin primer-kohteet.
kärki yksi: Miten löytää SNPs
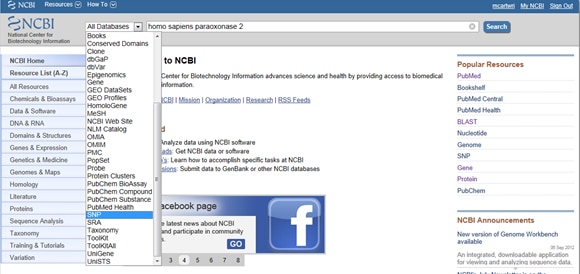
ottaen huomioon SNPs: n merkityksen sekä taudissa että tutkimuksessa, NCBI tarjoaa työkaluja geenin raportoidun SNPs: n kokoamiseen. Jos haluat löytää SNPs: n, aloita NCBI: n kotisivulta ja kirjoita kiinnostavan geenisi hakupalkkiin. Valitse SNP Kaikki tietokannat-pudotusvalikosta hakupalkin vasemmalta puolelta, kuten alla on esitetty:
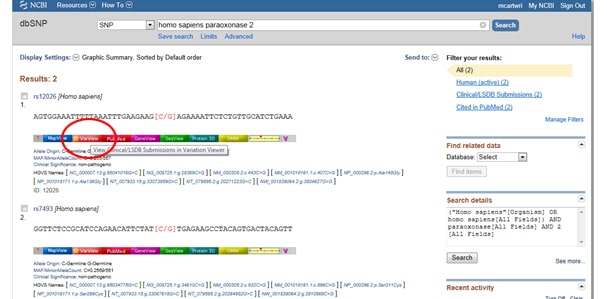
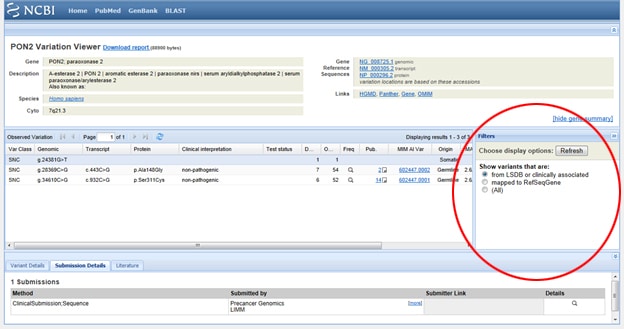
temppu yksi: pitääkö tulokset suodattaa niin, että tarkastellaan vain kliinisesti liittyviä tuloksia? Siirry näyttöruutuun, jossa on Suodattimet SNP-listan oikealla puolella havaitun vaihtelun kohdalla. Kun olet valinnut suodatinvaihtoehdon, muista painaa Päivitä-painiketta.
Tip Two: kuinka suunnitella alukkeet
NCBI tarjoaa Primer-Blastin alukkeiden automaattiseen suunnitteluun kyselysekvenssin perusteella. Jos haluat aloittaa primereiden suunnittelun, siirry BLAST-kotisivulle ja selaa Primer-BLAST-vaihtoehtoa Specialized BLAST-kohdassa. Syötä kohdesekvenssi joko cut-and-paste tai, jos se on NCBI: n tietokannoissa, liittymisnumerona. Kattaa joitakin muokkausvaihtoehtoja alla, mutta tässä vaiheessa, voit luoda alukkeet tekemättä mitään ylimääräisiä räätälöinnin!
alue: sekvenssin syöttämistä koskevan ruudun oikealla puolella voit määrittää kohteen tarkan alueen (numeroituna 5: stä 3: een, sekvenssin alusta alkaen), jota harkitaan eteenpäin ja taaksepäin suuntautuvien alukkeiden suunnittelussa.
Käytä omaa forward primeria (5′->3′ plus Strandilla): Valitse tämä, Jos olet jo suunnitellut alukkeesi ja haluat Primer-Blastin tarjoavan niistä Analytiikkaa (esim.Tm).
PCR-tuotteiden koko: Aseta PCR-tuotteiden hyväksyttävien pituuksien vaihteluväli tässä.
# of primers to return: tämä asettaa haluamasi määrän ehdokkaita primers-sarjoja harkittavaksi. Huomaa, että se ei ole takuu, varsinkin jos parametrit ovat liian tiukat tai järjettömät (esim.määritit tuotteen alle PCR tuotekoko, joka ei voi olla yli 500 bp, mutta alle alue haluat vain harkita alukkeet yli 1 kb välein).
pohjamaalien sulamislämpötilat: Tämän avulla voit määrittää TM: n (saadaksesi nopean kertauksen sulamislämpötilasta, tutustu vinkkeihimme qPCR: ään ja säännölliseen PCR primer-suunnitteluun).
Eksoniliitosspanos: jos haluat sulkea pois genomisen DNA: n (jossa eksonit jaetaan ei-koodaavilla introneilla), aseta tämä Primer must span ekson-ekson-liitosspanos.
spesifisyyden tarkistus: ellet halua Primer-Blastin palauttavan alukkeita, jotka menevät pois kohteesta (ei yleensä suositella!), jätä tämä tarkistettu ja määrittää organismin näytteesi tulevat sekä mitä tietokantaa käyttää, riippuen siitä, jos olet kohteena mRNA, gDNA, jne. Sallimalla spesifisyyden tarkistuksen, – Primer-BLAST sulkee pois alukkeet, jotka voisivat vahvistaa jotain kohdesekvenssin ulkopuolella.
Liitosvaihtoehdon käsittely: Jos valitset tämän vaihtoehdon – se on mahdollista vain, jos työstät mRNA – sekvenssejä-Primer-BLAST ei sulje pois primer-BLAST-pohjustuspareja, jotka voivat vahvistaa Useita mRNA-liitosvariantteja kohteestasi. Tämä ei kuitenkaan tarkoita, että se antaa sinulle pohjustuspareja, jotka kattavat kaikki tunnetut liitosvaihtoehdot! Löysäät vain tavoitekriteerejäsi.
kun olet syöttänyt sekvenssisi ja muokannut sitä tarpeen mukaan, vieritä sivun alareunaan ja käytä tarkistamisen jälkeen uutta graafista näkymää, paina Get Primers. Tämä palauttaa kartan siitä, missä ehdotetut pohjustusparit vahvistavat tavoitettasi, sekä analyysit alukkeista: niiden pituus, tarkka sijainti, vastaavat Tm: n, GC% ja pisteet, jotka heijastavat itsensä täydentävyyttä (0,00 heijastaen ennustettua täydentävyyttä).
vinkki kolme: miten ennakoida Aluketavoitteet
miten voi tarkistaa, osuvatko alukkeesi johonkin off-Targetiin? Aloita räjäytys. Syötä kyselyruutuun eteenpäin suuntautuva primer (5: stä 3: een). Kirjoita nyt 20 N: n riviin erottaaksesi alukkeet yksittäisiin, ei-päällekkäisiin linjauksiin. N: n jälkeen kirjoita Käänteinen primer (myös 5′ to 3′), kuten alla on esitetty:
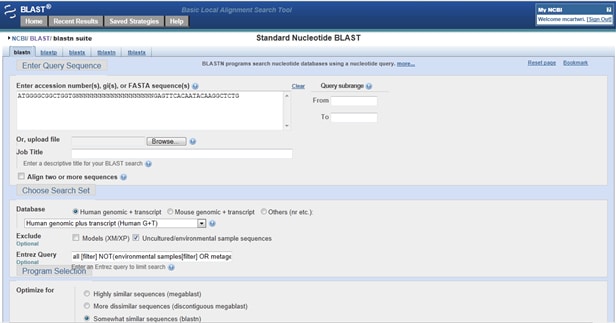
kun saat tuloksesi, tarkista ne tiettyjen yhdistelmien varalta. Jos forward primer asettuu eteen strand (huomautus Strand Plus / Plus) ja käänteinen primer kohdistaa saman osuma, mutta käänteinen strand (Strand Plus / miinus), niin alukkeet voivat vahvistaa, että osuma.
temppu kaksi: sisältävätkö tuloksesi asioita, jotka eivät todennäköisesti ole saastuttaneet PCR-näytteitäsi, kuten oliivipaviaaneja ja neandertalilaisia? Jos käytät ihmisen tai hiiren näytteitä, varmista, että sinulla on ne, jotka on määritelty tietokannassa. Vaihtoehtoisesti voit sulkea pois tietyt lajit.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
onko tämä auttanut sinua? Jaa sitten verkkosi kanssa.