den grundlæggende algoritme til søgning efter lokal tilpasning (BLAST) er kernen i en gratis pakke med online ressourcer, der er tilgængelige via National Center for Biotechnology Information (NCBI). Mens de fleste forskere er opmærksomme på BLAST som et sekvensjusteringsværktøj, tilbyder NCBI ‘ s BLAST suite så meget mere! Jeg vil dække dybtgående, hvordan man bruger disse ressourcer til at lokalisere enkelt nukleotidpolymorfier (SNP ‘ er) i et gen; design primere med Primer-BLAST; og validere primer mål.
Tip en: Sådan finder du SNP ‘er
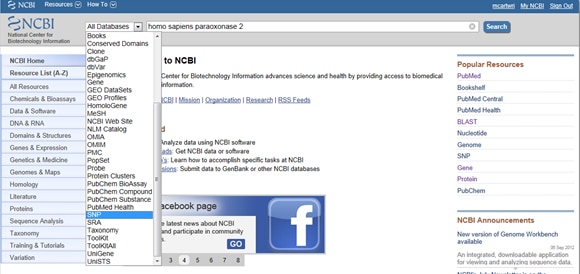
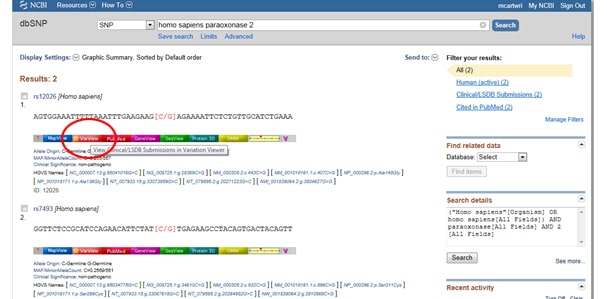
i betragtning af vigtigheden af SNP’ er i både sygdom og forskning giver NCBI værktøjer til at samle et gens rapporterede SNP ‘ er. For at finde SNP ‘er skal du starte på NCBI’ s hjemmeside og skrive dit gen af interesse i søgefeltet. Vælg SNP fra rullemenuen Alle databaser til venstre for søgefeltet, som vist nedenfor:
Tip to: Sådan designes primere
NCBI giver Primer-BLAST til automatisk at designe primere baseret på en forespørgselssekvens. For at begynde at designe primere skal du gå til BLAST-hjemmesiden og rulle ned til Primer-BLAST-indstillingen under specialiseret BLAST. Indtast din målsekvens enten ved at klippe og indsætte eller, hvis den er angivet i NCBI ‘ s databaser, som et tiltrædelsesnummer. Jeg dækker nogle tilpasningsmuligheder nedenfor, men på dette tidspunkt kan du generere primere uden at foretage yderligere tilpasning!
rækkevidde: til højre for boksen til indtastning af din sekvens kan du angive det nøjagtige interval (som nummereret 5′ til 3′ fra starten af din sekvens) for det mål, der vil blive overvejet til design af primere fremad og bagud.
brug min egen fremadrettede primer (5 ‘ – >3’ på plus strand): Vælg dette, hvis du allerede har designet dine primere og ønsker, at Primer-BLAST skal give nogle analyser (f.eks.
PCR-produktstørrelse: Indstil området for acceptable længder af PCR-produkterne her.
# af primere, der skal returneres: dette indstiller dit foretrukne antal kandidatsæt af primere, der skal overvejes. Bemærk, at det ikke er en garanti, især hvis dine parametre er for strenge eller meningsløse (f.eks. du har angivet et produkt under PCR-produktstørrelse, der ikke kan være mere end 500 bp, men under rækkevidde vil du kun overveje primere mere end 1 kb fra hinanden).
Primer smeltetemperatur: Dette giver dig mulighed for at specificere din Tm (for en hurtig genopfriskning af smeltetemperaturen, se vores tip til kpcr og almindeligt PCR-primerdesign).hvis du vil udelukke genomisk DNA (hvor eksoner er divideret med ikke-kodende introner), skal du indstille dette til Primer skal spænde over et ekson-ekson-kryds.
Specificitetskontrol: medmindre du vil have Primer-BLAST til at returnere primere, der vil gå off-target (anbefales generelt ikke!), lad dette kontrolleres og angiv den organisme, dine prøver kommer fra, samt hvilken Database der skal bruges, afhængigt af om du målretter mod mRNA, gDNA osv. Ved at aktivere specificitetskontrollen udelukker Primer-BLAST primere, der kan forstærke noget uden for din målsekvens.
Splice variant håndtering: hvis du vælger denne indstilling – kun muligt, hvis du arbejder ud af mRNA sekvenser – så Primer-BLAST vil ikke udelukke primer par, der kunne forstærke flere mRNA splice varianter af dit mål. Dette betyder dog ikke, at det vil give dig primerpar, der omfatter alle kendte splejsningsvarianter! Du løsner simpelthen dine målkriterier.
Når du har indtastet din sekvens og tilpasset efter behov, skal du rulle ned til bunden af siden, og efter at have kontrolleret Brug ny grafisk visning, skal du trykke på få primere. Dette vil returnere et kort over, hvor de foreslåede primerpar vil forstærke dit mål, samt analyser på primerne: deres længde, præcise placering, respektive Tm ‘ er, GC% og scoringer, der afspejler selvkomplementaritet (med 0,00 afspejler ingen forudsagt komplementering).
Tip tre: Sådan forudsiger du Primermål
hvordan kan du kontrollere, om dine primere rammer noget uden for målet? Gå til Primer-BLAST. I forespørgselsfeltet skal du indtaste din fremadgående primer (5′ til 3′). Skriv nu 20 N ‘ er i træk for at adskille primerne i individuelle, ikke-overlappende justeringer. Efter N’ erne skal du indtaste din omvendte primer (også 5′ til 3′) som vist nedenfor:
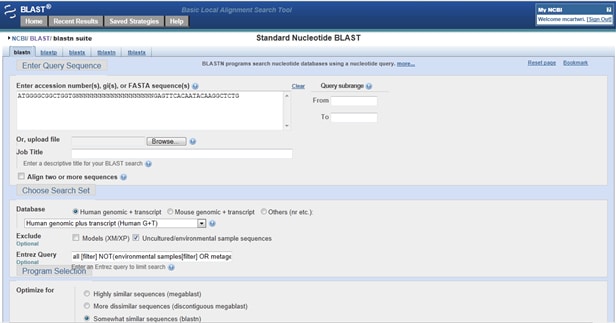
når du har fået dine resultater, skal du kontrollere dem for bestemte kombinationer. Hvis din fremadgående primer justeres på den forreste streng (kommenteret streng Plus/Plus), og din omvendte primer justeres til det samme hit, men på den omvendte streng (streng Plus/Minus), kan dine primere forstærke det hit.Trick to: inkluderer dine resultater ting, der sandsynligvis ikke har forurenet dine PCR-prøver, såsom olivenbavianer og neandertalere? Hvis du arbejder med prøver fra mennesker eller mus, skal du sørge for at have dem, der er angivet under Database. Alternativt kan du udelukke bestemte arter.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
har dette hjulpet dig? Så del venligst med dit netværk.