Základní Local Alignment Search Tool (BLAST) algoritmus je v srdci zdarma sadu on-line zdrojů k dispozici prostřednictvím Národního Centra pro Biotechnologické Informace (NCBI). Zatímco většina vědců si je vědoma BLAST jako nástroje pro zarovnání sekvencí, NCBI BLAST suite nabízí mnohem více! Budu krýt v-hloubky, jak používat tyto zdroje k vyhledání jednonukleotidových polymorfismů (Snp) v genu; design primerů s Primer-BLAST; a ověřit primer cíle.
Tip jedna: Jak najít SNP
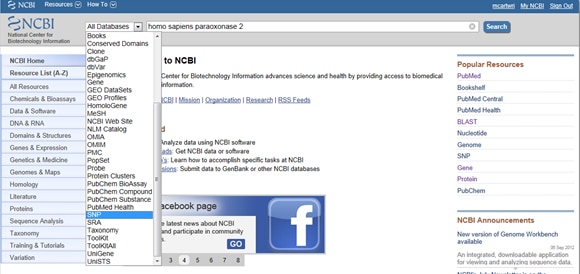
vzhledem k významu SNP jak v nemoci, tak ve výzkumu poskytuje NCBI nástroje pro shromažďování hlášených SNP genu. Chcete-li najít SNP, začněte na domovské stránce NCBI a do vyhledávacího pole zadejte svůj Gen zájmu. Vyberte SNP ze Všech Databází rozbalovací menu na levé straně vyhledávacího řádku, jak je uvedeno níže:
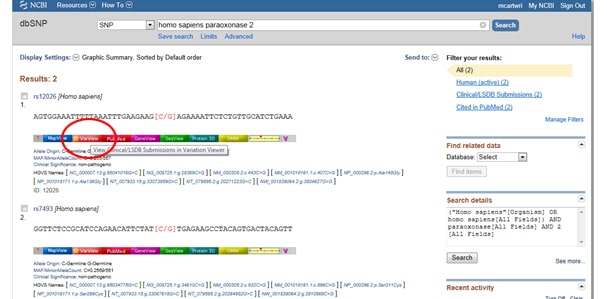
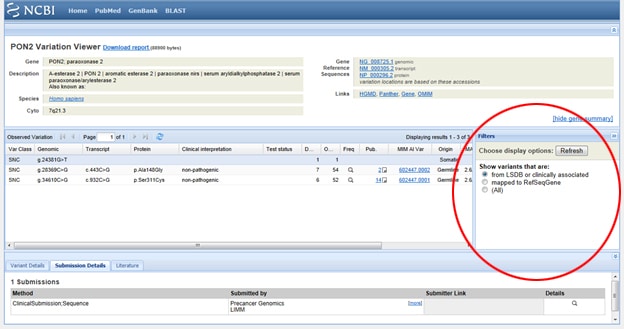
Trick One: potřebujete filtrovat výsledky tak, abyste se dívali pouze na klinicky související výsledky? Přejděte do tohoto zobrazovacího pole označeného filtry napravo od Seznamu SNP v části pozorovaná Změna. Po výběru možnosti filtru nezapomeňte stisknout tlačítko Obnovit.
Tip Dvě: Jak Navrhnout Primery
NCBI poskytuje Primer-BLAST pro automatické navrhování primerů na základě dotazu sekvence. Chcete-li začít navrhovat základní nátěry, přejděte na domovskou stránku BLAST a přejděte dolů na možnost Primer-BLAST pod Specialized BLAST. Zadejte svou cílovou sekvenci buď vyjmutím a vložením, nebo, pokud je uvedena v databázích NCBI, jako přístupové číslo. Níže uvádím některé možnosti přizpůsobení, ale v tomto okamžiku můžete vytvářet základní nátěry bez dalšího přizpůsobení!
rozsah: v pravé části pole pro zadání sekvence můžete zadat přesný rozsah (očíslovaný 5′ až 3′ od začátku sekvence) cíle, který bude zvažován pro návrh primerů vpřed a vzad.
Použít vlastní forward primer (5′->3′ na plus pramen): Zvolte tuto možnost, pokud jste již navrhli primery a chci Primer-BLAST poskytnout některé analytické (např. Tm) o nich.
Velikost produktu PCR: Zde nastavte rozsah přijatelných délek produktů PCR.
# primerů k návratu: tím se nastaví preferovaný počet kandidátských sad primerů, které je třeba zvážit. Všimněte si, že to není záruka, a to zejména pokud vaše parametry jsou příliš přísné a nesmyslné (např. zadaného výrobku podle PCR produkt velikosti, že nemůže být větší než 500 bp, ale podle Rozsahu pouze chtít, aby zvážila primery více než 1 kb od sebe).
teploty tání primeru: To vám umožní určit Tm (pro rychlé obnovení teploty tání, podívejte se na naše tipy pro qPCR a pravidelné PCR primer design).
Exon junction span: Pokud chcete vyloučit genomické DNA (kde exons jsou rozděleny podle nekódující introny), pak nastavte Primer musí span exon-exon junction.
kontrola specifičnosti: pokud nechcete, aby Primer-BLAST vrátil primery, které půjdou mimo cíl (obecně se nedoporučuje!), nechte to zkontrolovat a určete organismus, ze kterého vaše vzorky pocházejí, a také databázi, kterou chcete použít, v závislosti na tom, zda cílíte na mRNA, gDNA atd. Povolením kontroly specifičnosti Primer-BLAST vyloučí primery, které by mohly zesílit něco mimo vaši cílovou sekvenci.
Splice varianta manipulace: Pokud vyberete tuto možnost – možné pouze pokud pracujete z mRNA sekvence – a pak Primer-BLAST nebude vylučovat páry primerů, které by mohly zesílit více mRNA sestřihu varianty svého cíle. To však neznamená, že vám poskytne páry primerů, které zahrnují všechny známé varianty spojů! Jednoduše uvolňujete Cílová kritéria.
Jakmile jste zadali vaše sekvence a upravit podle potřeby, přejděte dolů do spodní části stránky a po kontrole Používat nové grafické zobrazení, zásah Se Primery. To se vrátí mapu, kde navrhl primer párů bude zesilovat svůj cíl, stejně jako analytics na primery: jejich délka, přesné umístění, příslušné Tm, GC%, a skóre odrážející self-doplňkovost (s 0.00 odráží č. předpověděl doplnění).
Tip Tři: jak předpovědět cíle primeru
jak můžete zkontrolovat, zda vaše primery zasáhly něco mimo cíl? Přejít na Primer-BLAST. Do pole dotazu zadejte dopředný primer (5 ‚až 3′). Nyní zadejte 20 N v řadě, abyste rozdělili primery na jednotlivé, nepřekrývající se zarovnání. Po N, zadejte své reverse primer (5′ k 3‘), jak je uvedeno níže:
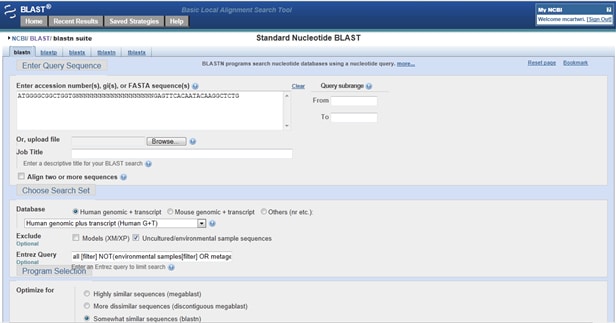
jakmile získáte výsledky, zkontrolujte je pro určité kombinace. Pokud váš forward primer zarovná na přední pramen (komentovaný Strand Plus/Plus) a reverzní primer zarovná na stejný hit, ale na zadní pramen (Pramen Plus/Minus), pak se vaše primery mohou zesilovat, že hit.
trik druhý: zahrnují vaše výsledky věci, které pravděpodobně kontaminovaly vaše vzorky PCR, jako jsou olivoví paviáni a neandrtálci? Pokud pracujete se vzorky lidí nebo myší, ujistěte se, že máte ty, které jsou uvedeny v databázi. Případně můžete vyloučit konkrétní druhy.
References and Additional Resources:
Blast Tips. 2007. NCBI. <http://www.ncbi.nlm.nih.gov/feed/rss.cgi?ChanKey=blasttips>
Frequently Asked Questions. NCBI BLAST Help. <http://www.ncbi.nlm.nih.gov/blast/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ>
Madden T. The BLAST Sequence Analysis Tool. 2003. <http://www.ncbi.nlm.nih.gov/books/NBK21097/>
Mount DW. Using the Basic Local Alignment Search Tool. 2004. Cold Spring Harbor Protocols. <http://cshprotocols.cshlp.org/content/2007/7/pdb.top17.full>
Wheeler D and Bhagwat M. BLAST QuickStart. 2007. Humana Press Inc. <http://www.ncbi.nlm.nih.gov/books/NBK1734/>
Má to pomohlo? Pak prosím sdílejte s vaší sítí.